Erilaiset visualisoinnit ja analyysiprosessi kokonaisuus
1 Kertausta
1.1 Datan käsittely
1.1.1 Taulukot
Yksinkertaiset taulukot
library(tidyverse)
sw <- dplyr::starwars
# yhden muuttujan luokkien frekvenssit
as.data.frame(
table(sw$species, useNA = "ifany")
)## Var1 Freq
## 1 Aleena 1
## 2 Besalisk 1
## 3 Cerean 1
## 4 Chagrian 1
## 5 Clawdite 1
## 6 Droid 5
## 7 Dug 1
## 8 Ewok 1
## 9 Geonosian 1
## 10 Gungan 3
## 11 Human 35
## 12 Hutt 1
## 13 Iktotchi 1
## 14 Kaleesh 1
## 15 Kaminoan 2
## 16 Kel Dor 1
## 17 Mirialan 2
## 18 Mon Calamari 1
## 19 Muun 1
## 20 Nautolan 1
## 21 Neimodian 1
## 22 Pau'an 1
## 23 Quermian 1
## 24 Rodian 1
## 25 Skakoan 1
## 26 Sullustan 1
## 27 Tholothian 1
## 28 Togruta 1
## 29 Toong 1
## 30 Toydarian 1
## 31 Trandoshan 1
## 32 Twi'lek 2
## 33 Vulptereen 1
## 34 Wookiee 2
## 35 Xexto 1
## 36 Yoda's species 1
## 37 Zabrak 2
## 38 <NA> 5# OR
sw %>% count(species)## # A tibble: 38 x 2
## species n
## <chr> <int>
## 1 Aleena 1
## 2 Besalisk 1
## 3 Cerean 1
## 4 Chagrian 1
## 5 Clawdite 1
## 6 Droid 5
## 7 Dug 1
## 8 Ewok 1
## 9 Geonosian 1
## 10 Gungan 3
## # ... with 28 more rows# yhden muuttujan luokkien osuudet
as.data.frame(
prop.table(table(sw$species, useNA = "ifany"))*100
)## Var1 Freq
## 1 Aleena 1.149425
## 2 Besalisk 1.149425
## 3 Cerean 1.149425
## 4 Chagrian 1.149425
## 5 Clawdite 1.149425
## 6 Droid 5.747126
## 7 Dug 1.149425
## 8 Ewok 1.149425
## 9 Geonosian 1.149425
## 10 Gungan 3.448276
## 11 Human 40.229885
## 12 Hutt 1.149425
## 13 Iktotchi 1.149425
## 14 Kaleesh 1.149425
## 15 Kaminoan 2.298851
## 16 Kel Dor 1.149425
## 17 Mirialan 2.298851
## 18 Mon Calamari 1.149425
## 19 Muun 1.149425
## 20 Nautolan 1.149425
## 21 Neimodian 1.149425
## 22 Pau'an 1.149425
## 23 Quermian 1.149425
## 24 Rodian 1.149425
## 25 Skakoan 1.149425
## 26 Sullustan 1.149425
## 27 Tholothian 1.149425
## 28 Togruta 1.149425
## 29 Toong 1.149425
## 30 Toydarian 1.149425
## 31 Trandoshan 1.149425
## 32 Twi'lek 2.298851
## 33 Vulptereen 1.149425
## 34 Wookiee 2.298851
## 35 Xexto 1.149425
## 36 Yoda's species 1.149425
## 37 Zabrak 2.298851
## 38 <NA> 5.747126# OR
sw %>% count(species) %>%
mutate(share = n/sum(n)*100)## # A tibble: 38 x 3
## species n share
## <chr> <int> <dbl>
## 1 Aleena 1 1.15
## 2 Besalisk 1 1.15
## 3 Cerean 1 1.15
## 4 Chagrian 1 1.15
## 5 Clawdite 1 1.15
## 6 Droid 5 5.75
## 7 Dug 1 1.15
## 8 Ewok 1 1.15
## 9 Geonosian 1 1.15
## 10 Gungan 3 3.45
## # ... with 28 more rows# kahden muuttujan luokkien frekvenssit
as.data.frame(
table(sw$species,sw$gender, useNA = "ifany")
)## Var1 Var2 Freq
## 1 Aleena female 0
## 2 Besalisk female 0
## 3 Cerean female 0
## 4 Chagrian female 0
## 5 Clawdite female 1
## 6 Droid female 0
## 7 Dug female 0
## 8 Ewok female 0
## 9 Geonosian female 0
## 10 Gungan female 0
## 11 Human female 9
## 12 Hutt female 0
## 13 Iktotchi female 0
## 14 Kaleesh female 0
## 15 Kaminoan female 1
## 16 Kel Dor female 0
## 17 Mirialan female 2
## 18 Mon Calamari female 0
## 19 Muun female 0
## 20 Nautolan female 0
## 21 Neimodian female 0
## 22 Pau'an female 0
## 23 Quermian female 0
## 24 Rodian female 0
## 25 Skakoan female 0
## 26 Sullustan female 0
## 27 Tholothian female 1
## 28 Togruta female 1
## 29 Toong female 0
## 30 Toydarian female 0
## 31 Trandoshan female 0
## 32 Twi'lek female 1
## 33 Vulptereen female 0
## 34 Wookiee female 0
## 35 Xexto female 0
## 36 Yoda's species female 0
## 37 Zabrak female 0
## 38 <NA> female 3
## 39 Aleena hermaphrodite 0
## 40 Besalisk hermaphrodite 0
## 41 Cerean hermaphrodite 0
## 42 Chagrian hermaphrodite 0
## 43 Clawdite hermaphrodite 0
## 44 Droid hermaphrodite 0
## 45 Dug hermaphrodite 0
## 46 Ewok hermaphrodite 0
## 47 Geonosian hermaphrodite 0
## 48 Gungan hermaphrodite 0
## 49 Human hermaphrodite 0
## 50 Hutt hermaphrodite 1
## 51 Iktotchi hermaphrodite 0
## 52 Kaleesh hermaphrodite 0
## 53 Kaminoan hermaphrodite 0
## 54 Kel Dor hermaphrodite 0
## 55 Mirialan hermaphrodite 0
## 56 Mon Calamari hermaphrodite 0
## 57 Muun hermaphrodite 0
## 58 Nautolan hermaphrodite 0
## 59 Neimodian hermaphrodite 0
## 60 Pau'an hermaphrodite 0
## 61 Quermian hermaphrodite 0
## 62 Rodian hermaphrodite 0
## 63 Skakoan hermaphrodite 0
## 64 Sullustan hermaphrodite 0
## 65 Tholothian hermaphrodite 0
## 66 Togruta hermaphrodite 0
## 67 Toong hermaphrodite 0
## 68 Toydarian hermaphrodite 0
## 69 Trandoshan hermaphrodite 0
## 70 Twi'lek hermaphrodite 0
## 71 Vulptereen hermaphrodite 0
## 72 Wookiee hermaphrodite 0
## 73 Xexto hermaphrodite 0
## 74 Yoda's species hermaphrodite 0
## 75 Zabrak hermaphrodite 0
## 76 <NA> hermaphrodite 0
## 77 Aleena male 1
## 78 Besalisk male 1
## 79 Cerean male 1
## 80 Chagrian male 1
## 81 Clawdite male 0
## 82 Droid male 0
## 83 Dug male 1
## 84 Ewok male 1
## 85 Geonosian male 1
## 86 Gungan male 3
## 87 Human male 26
## 88 Hutt male 0
## 89 Iktotchi male 1
## 90 Kaleesh male 1
## 91 Kaminoan male 1
## 92 Kel Dor male 1
## 93 Mirialan male 0
## 94 Mon Calamari male 1
## 95 Muun male 1
## 96 Nautolan male 1
## 97 Neimodian male 1
## 98 Pau'an male 1
## 99 Quermian male 1
## 100 Rodian male 1
## 101 Skakoan male 1
## 102 Sullustan male 1
## 103 Tholothian male 0
## 104 Togruta male 0
## 105 Toong male 1
## 106 Toydarian male 1
## 107 Trandoshan male 1
## 108 Twi'lek male 1
## 109 Vulptereen male 1
## 110 Wookiee male 2
## 111 Xexto male 1
## 112 Yoda's species male 1
## 113 Zabrak male 2
## 114 <NA> male 2
## 115 Aleena none 0
## 116 Besalisk none 0
## 117 Cerean none 0
## 118 Chagrian none 0
## 119 Clawdite none 0
## 120 Droid none 2
## 121 Dug none 0
## 122 Ewok none 0
## 123 Geonosian none 0
## 124 Gungan none 0
## 125 Human none 0
## 126 Hutt none 0
## 127 Iktotchi none 0
## 128 Kaleesh none 0
## 129 Kaminoan none 0
## 130 Kel Dor none 0
## 131 Mirialan none 0
## 132 Mon Calamari none 0
## 133 Muun none 0
## 134 Nautolan none 0
## 135 Neimodian none 0
## 136 Pau'an none 0
## 137 Quermian none 0
## 138 Rodian none 0
## 139 Skakoan none 0
## 140 Sullustan none 0
## 141 Tholothian none 0
## 142 Togruta none 0
## 143 Toong none 0
## 144 Toydarian none 0
## 145 Trandoshan none 0
## 146 Twi'lek none 0
## 147 Vulptereen none 0
## 148 Wookiee none 0
## 149 Xexto none 0
## 150 Yoda's species none 0
## 151 Zabrak none 0
## 152 <NA> none 0
## 153 Aleena <NA> 0
## 154 Besalisk <NA> 0
## 155 Cerean <NA> 0
## 156 Chagrian <NA> 0
## 157 Clawdite <NA> 0
## 158 Droid <NA> 3
## 159 Dug <NA> 0
## 160 Ewok <NA> 0
## 161 Geonosian <NA> 0
## 162 Gungan <NA> 0
## 163 Human <NA> 0
## 164 Hutt <NA> 0
## 165 Iktotchi <NA> 0
## 166 Kaleesh <NA> 0
## 167 Kaminoan <NA> 0
## 168 Kel Dor <NA> 0
## 169 Mirialan <NA> 0
## 170 Mon Calamari <NA> 0
## 171 Muun <NA> 0
## 172 Nautolan <NA> 0
## 173 Neimodian <NA> 0
## 174 Pau'an <NA> 0
## 175 Quermian <NA> 0
## 176 Rodian <NA> 0
## 177 Skakoan <NA> 0
## 178 Sullustan <NA> 0
## 179 Tholothian <NA> 0
## 180 Togruta <NA> 0
## 181 Toong <NA> 0
## 182 Toydarian <NA> 0
## 183 Trandoshan <NA> 0
## 184 Twi'lek <NA> 0
## 185 Vulptereen <NA> 0
## 186 Wookiee <NA> 0
## 187 Xexto <NA> 0
## 188 Yoda's species <NA> 0
## 189 Zabrak <NA> 0
## 190 <NA> <NA> 0# Or
sw %>%
group_by(species) %>%
count(gender)## # A tibble: 43 x 3
## # Groups: species [38]
## species gender n
## <chr> <chr> <int>
## 1 Aleena male 1
## 2 Besalisk male 1
## 3 Cerean male 1
## 4 Chagrian male 1
## 5 Clawdite female 1
## 6 Droid none 2
## 7 Droid <NA> 3
## 8 Dug male 1
## 9 Ewok male 1
## 10 Geonosian male 1
## # ... with 33 more rows# kahden muuttujan luokkien frekvenssit
prop.table(table(sw$species,sw$gender))##
## female hermaphrodite male none
## Aleena 0.00000000 0.00000000 0.01265823 0.00000000
## Besalisk 0.00000000 0.00000000 0.01265823 0.00000000
## Cerean 0.00000000 0.00000000 0.01265823 0.00000000
## Chagrian 0.00000000 0.00000000 0.01265823 0.00000000
## Clawdite 0.01265823 0.00000000 0.00000000 0.00000000
## Droid 0.00000000 0.00000000 0.00000000 0.02531646
## Dug 0.00000000 0.00000000 0.01265823 0.00000000
## Ewok 0.00000000 0.00000000 0.01265823 0.00000000
## Geonosian 0.00000000 0.00000000 0.01265823 0.00000000
## Gungan 0.00000000 0.00000000 0.03797468 0.00000000
## Human 0.11392405 0.00000000 0.32911392 0.00000000
## Hutt 0.00000000 0.01265823 0.00000000 0.00000000
## Iktotchi 0.00000000 0.00000000 0.01265823 0.00000000
## Kaleesh 0.00000000 0.00000000 0.01265823 0.00000000
## Kaminoan 0.01265823 0.00000000 0.01265823 0.00000000
## Kel Dor 0.00000000 0.00000000 0.01265823 0.00000000
## Mirialan 0.02531646 0.00000000 0.00000000 0.00000000
## Mon Calamari 0.00000000 0.00000000 0.01265823 0.00000000
## Muun 0.00000000 0.00000000 0.01265823 0.00000000
## Nautolan 0.00000000 0.00000000 0.01265823 0.00000000
## Neimodian 0.00000000 0.00000000 0.01265823 0.00000000
## Pau'an 0.00000000 0.00000000 0.01265823 0.00000000
## Quermian 0.00000000 0.00000000 0.01265823 0.00000000
## Rodian 0.00000000 0.00000000 0.01265823 0.00000000
## Skakoan 0.00000000 0.00000000 0.01265823 0.00000000
## Sullustan 0.00000000 0.00000000 0.01265823 0.00000000
## Tholothian 0.01265823 0.00000000 0.00000000 0.00000000
## Togruta 0.01265823 0.00000000 0.00000000 0.00000000
## Toong 0.00000000 0.00000000 0.01265823 0.00000000
## Toydarian 0.00000000 0.00000000 0.01265823 0.00000000
## Trandoshan 0.00000000 0.00000000 0.01265823 0.00000000
## Twi'lek 0.01265823 0.00000000 0.01265823 0.00000000
## Vulptereen 0.00000000 0.00000000 0.01265823 0.00000000
## Wookiee 0.00000000 0.00000000 0.02531646 0.00000000
## Xexto 0.00000000 0.00000000 0.01265823 0.00000000
## Yoda's species 0.00000000 0.00000000 0.01265823 0.00000000
## Zabrak 0.00000000 0.00000000 0.02531646 0.00000000prop.table(table(sw$species,sw$gender),1)##
## female hermaphrodite male none
## Aleena 0.0000000 0.0000000 1.0000000 0.0000000
## Besalisk 0.0000000 0.0000000 1.0000000 0.0000000
## Cerean 0.0000000 0.0000000 1.0000000 0.0000000
## Chagrian 0.0000000 0.0000000 1.0000000 0.0000000
## Clawdite 1.0000000 0.0000000 0.0000000 0.0000000
## Droid 0.0000000 0.0000000 0.0000000 1.0000000
## Dug 0.0000000 0.0000000 1.0000000 0.0000000
## Ewok 0.0000000 0.0000000 1.0000000 0.0000000
## Geonosian 0.0000000 0.0000000 1.0000000 0.0000000
## Gungan 0.0000000 0.0000000 1.0000000 0.0000000
## Human 0.2571429 0.0000000 0.7428571 0.0000000
## Hutt 0.0000000 1.0000000 0.0000000 0.0000000
## Iktotchi 0.0000000 0.0000000 1.0000000 0.0000000
## Kaleesh 0.0000000 0.0000000 1.0000000 0.0000000
## Kaminoan 0.5000000 0.0000000 0.5000000 0.0000000
## Kel Dor 0.0000000 0.0000000 1.0000000 0.0000000
## Mirialan 1.0000000 0.0000000 0.0000000 0.0000000
## Mon Calamari 0.0000000 0.0000000 1.0000000 0.0000000
## Muun 0.0000000 0.0000000 1.0000000 0.0000000
## Nautolan 0.0000000 0.0000000 1.0000000 0.0000000
## Neimodian 0.0000000 0.0000000 1.0000000 0.0000000
## Pau'an 0.0000000 0.0000000 1.0000000 0.0000000
## Quermian 0.0000000 0.0000000 1.0000000 0.0000000
## Rodian 0.0000000 0.0000000 1.0000000 0.0000000
## Skakoan 0.0000000 0.0000000 1.0000000 0.0000000
## Sullustan 0.0000000 0.0000000 1.0000000 0.0000000
## Tholothian 1.0000000 0.0000000 0.0000000 0.0000000
## Togruta 1.0000000 0.0000000 0.0000000 0.0000000
## Toong 0.0000000 0.0000000 1.0000000 0.0000000
## Toydarian 0.0000000 0.0000000 1.0000000 0.0000000
## Trandoshan 0.0000000 0.0000000 1.0000000 0.0000000
## Twi'lek 0.5000000 0.0000000 0.5000000 0.0000000
## Vulptereen 0.0000000 0.0000000 1.0000000 0.0000000
## Wookiee 0.0000000 0.0000000 1.0000000 0.0000000
## Xexto 0.0000000 0.0000000 1.0000000 0.0000000
## Yoda's species 0.0000000 0.0000000 1.0000000 0.0000000
## Zabrak 0.0000000 0.0000000 1.0000000 0.0000000prop.table(table(sw$species,sw$gender),2)##
## female hermaphrodite male none
## Aleena 0.00000000 0.00000000 0.01666667 0.00000000
## Besalisk 0.00000000 0.00000000 0.01666667 0.00000000
## Cerean 0.00000000 0.00000000 0.01666667 0.00000000
## Chagrian 0.00000000 0.00000000 0.01666667 0.00000000
## Clawdite 0.06250000 0.00000000 0.00000000 0.00000000
## Droid 0.00000000 0.00000000 0.00000000 1.00000000
## Dug 0.00000000 0.00000000 0.01666667 0.00000000
## Ewok 0.00000000 0.00000000 0.01666667 0.00000000
## Geonosian 0.00000000 0.00000000 0.01666667 0.00000000
## Gungan 0.00000000 0.00000000 0.05000000 0.00000000
## Human 0.56250000 0.00000000 0.43333333 0.00000000
## Hutt 0.00000000 1.00000000 0.00000000 0.00000000
## Iktotchi 0.00000000 0.00000000 0.01666667 0.00000000
## Kaleesh 0.00000000 0.00000000 0.01666667 0.00000000
## Kaminoan 0.06250000 0.00000000 0.01666667 0.00000000
## Kel Dor 0.00000000 0.00000000 0.01666667 0.00000000
## Mirialan 0.12500000 0.00000000 0.00000000 0.00000000
## Mon Calamari 0.00000000 0.00000000 0.01666667 0.00000000
## Muun 0.00000000 0.00000000 0.01666667 0.00000000
## Nautolan 0.00000000 0.00000000 0.01666667 0.00000000
## Neimodian 0.00000000 0.00000000 0.01666667 0.00000000
## Pau'an 0.00000000 0.00000000 0.01666667 0.00000000
## Quermian 0.00000000 0.00000000 0.01666667 0.00000000
## Rodian 0.00000000 0.00000000 0.01666667 0.00000000
## Skakoan 0.00000000 0.00000000 0.01666667 0.00000000
## Sullustan 0.00000000 0.00000000 0.01666667 0.00000000
## Tholothian 0.06250000 0.00000000 0.00000000 0.00000000
## Togruta 0.06250000 0.00000000 0.00000000 0.00000000
## Toong 0.00000000 0.00000000 0.01666667 0.00000000
## Toydarian 0.00000000 0.00000000 0.01666667 0.00000000
## Trandoshan 0.00000000 0.00000000 0.01666667 0.00000000
## Twi'lek 0.06250000 0.00000000 0.01666667 0.00000000
## Vulptereen 0.00000000 0.00000000 0.01666667 0.00000000
## Wookiee 0.00000000 0.00000000 0.03333333 0.00000000
## Xexto 0.00000000 0.00000000 0.01666667 0.00000000
## Yoda's species 0.00000000 0.00000000 0.01666667 0.00000000
## Zabrak 0.00000000 0.00000000 0.03333333 0.000000001.1.2 Muuttujien uudelleenluokittelu
# Jatkuvan pilkkominen luokkiin
sw$pituus_lk[sw$height < 100] <- "alle metrin"
sw$pituus_lk[sw$height >= 100 & sw$height < 150] <- "metristä puoleentoista"
sw$pituus_lk[sw$height >= 150 & sw$height < 200] <- "puolestatoista kahteen metriin"
sw$pituus_lk[sw$height >= 200] <- "yli kaksi metriä"
table(sw$pituus_lk, useNA = "ifany")##
## alle metrin metristä puoleentoista
## 7 3
## puolestatoista kahteen metriin yli kaksi metriä
## 60 11
## <NA>
## 6sw <- sw %>%
mutate(height_lk = case_when(
height < 100 ~ "alle metrin",
height >= 100 & height < 150 ~ "metristä puoleentoista",
height >= 150 & height < 200 ~ "puolestatoista kahteen metriin",
height >= 200 ~ "yli kaksi metriä",
TRUE ~ "ei pituutta datassa"
))
sw %>% count(height_lk)## # A tibble: 5 x 2
## height_lk n
## <chr> <int>
## 1 alle metrin 7
## 2 ei pituutta datassa 6
## 3 metristä puoleentoista 3
## 4 puolestatoista kahteen metriin 60
## 5 yli kaksi metriä 111.1.3 Säännölliset lausekkeet eli regular expressions
- http://stat545.com/block022_regular-expression.html
- Introduction to stringr https://cran.r-project.org/web/packages/stringr/vignettes/stringr.html
library(tidyverse)
## ------------------------------------------------------------------------
sw <- dplyr::starwars
# str(sw)
# summary(sw)
## ------------------------------------------------------------------------
head(sw)## # A tibble: 6 x 13
## name height mass hair_color skin_color eye_color birth_year gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
## 1 Luke Sky… 172 77. blond fair blue 19.0 male
## 2 C-3PO 167 75. <NA> gold yellow 112. <NA>
## 3 R2-D2 96 32. <NA> white, bl… red 33.0 <NA>
## 4 Darth Va… 202 136. none white yellow 41.9 male
## 5 Leia Org… 150 49. brown light brown 19.0 female
## 6 Owen Lars 178 120. brown, gr… light blue 52.0 male
## # ... with 5 more variables: homeworld <chr>, species <chr>, films <list>,
## # vehicles <list>, starships <list>## ------------------------------------------------------------------------
sw %>% filter(species == "Human")## # A tibble: 35 x 13
## name height mass hair_color skin_color eye_color birth_year gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
## 1 Luke Sk… 172 77. blond fair blue 19.0 male
## 2 Darth V… 202 136. none white yellow 41.9 male
## 3 Leia Or… 150 49. brown light brown 19.0 female
## 4 Owen La… 178 120. brown, gr… light blue 52.0 male
## 5 Beru Wh… 165 75. brown light blue 47.0 female
## 6 Biggs D… 183 84. black light brown 24.0 male
## 7 Obi-Wan… 182 77. auburn, w… fair blue-gray 57.0 male
## 8 Anakin … 188 84. blond fair blue 41.9 male
## 9 Wilhuff… 180 NA auburn, g… fair blue 64.0 male
## 10 Han Solo 180 80. brown fair brown 29.0 male
## # ... with 25 more rows, and 5 more variables: homeworld <chr>,
## # species <chr>, films <list>, vehicles <list>, starships <list>## ------------------------------------------------------------------------
sw %>% filter(species %in% c("Human","Droid"))## # A tibble: 40 x 13
## name height mass hair_color skin_color eye_color birth_year gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
## 1 Luke Sk… 172 77. blond fair blue 19.0 male
## 2 C-3PO 167 75. <NA> gold yellow 112. <NA>
## 3 R2-D2 96 32. <NA> white, bl… red 33.0 <NA>
## 4 Darth V… 202 136. none white yellow 41.9 male
## 5 Leia Or… 150 49. brown light brown 19.0 female
## 6 Owen La… 178 120. brown, gr… light blue 52.0 male
## 7 Beru Wh… 165 75. brown light blue 47.0 female
## 8 R5-D4 97 32. <NA> white, red red NA <NA>
## 9 Biggs D… 183 84. black light brown 24.0 male
## 10 Obi-Wan… 182 77. auburn, w… fair blue-gray 57.0 male
## # ... with 30 more rows, and 5 more variables: homeworld <chr>,
## # species <chr>, films <list>, vehicles <list>, starships <list>## ------------------------------------------------------------------------
rodut <- c("Human","Droid","Wookiee","Rodian","Hutt","Yoda's species","Trandoshan",
"Mon Calamari","Ewok","Sullustan","Neimodian","Gungan","NA","Toydarian",
"Dug","Zabrak","Twi'lek","Vulptereen","Xexto","Toong","Cerean","Nautolan",
"Tholothian","Iktotchi","Quermian","Kel Dor","Chagrian","Geonosian","Mirialan",
"Clawdite","Besalisk","Kaminoan","Aleena","Skakoan","Muun","Togruta","Kaleesh","Pau'an")
grep("n", rodut) # indeksit## [1] 1 4 7 8 10 11 12 14 18 20 21 22 23 25 27 28 29 32 33 34 35 38grep("n", rodut, value=TRUE) # arvot## [1] "Human" "Rodian" "Trandoshan" "Mon Calamari"
## [5] "Sullustan" "Neimodian" "Gungan" "Toydarian"
## [9] "Vulptereen" "Toong" "Cerean" "Nautolan"
## [13] "Tholothian" "Quermian" "Chagrian" "Geonosian"
## [17] "Mirialan" "Kaminoan" "Aleena" "Skakoan"
## [21] "Muun" "Pau'an"## ------------------------------------------------------------------------
grep("k$", rodut, value=TRUE)## [1] "Ewok" "Zabrak" "Twi'lek" "Besalisk"## ------------------------------------------------------------------------
grepl("k$", rodut)## [1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE
## [12] FALSE FALSE FALSE FALSE TRUE TRUE FALSE FALSE FALSE FALSE FALSE
## [23] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE
## [34] FALSE FALSE FALSE FALSE FALSE# filtteroi rivit regular expressioilla
sw[grepl("k$", sw$species),c("name","species")]## # A tibble: 6 x 2
## name species
## <chr> <chr>
## 1 Wicket Systri Warrick Ewok
## 2 Darth Maul Zabrak
## 3 Bib Fortuna Twi'lek
## 4 Ayla Secura Twi'lek
## 5 Eeth Koth Zabrak
## 6 Dexter Jettster Besalisk# Or
sw %>% filter(grepl("k$", species)) %>% select(name,species)## # A tibble: 6 x 2
## name species
## <chr> <chr>
## 1 Wicket Systri Warrick Ewok
## 2 Darth Maul Zabrak
## 3 Bib Fortuna Twi'lek
## 4 Ayla Secura Twi'lek
## 5 Eeth Koth Zabrak
## 6 Dexter Jettster Besalisk## ------------------------------------------------------------------------
grep("^hair", names(sw), value=TRUE)## [1] "hair_color"makenoise <- function(n, selection = letters, length = 5){
apply(replicate(n, sample(c(selection, ""), length, replace=TRUE, prob=c(rep(0.7/length(selection), length(selection)), .3))), 2, paste0, collapse="")
}
n <- 10
noisystring <- paste0(makenoise(n), sample(1990:2015, n, rep=T), makenoise(n))
## ------------------------------------------------------------------------
noisystring## [1] "rei1996lpe" "zxgf2000ijhy" "knglb2008isxqp" "fk1995pbr"
## [5] "tgh1998pmpd" "e2014tebyd" "ku1998ifxa" "nboc2004qtngg"
## [9] "vzrc2013hpc" "kam2007lb"## ------------------------------------------------------------------------
gsub("[a-zA-Z]", "", noisystring)## [1] "1996" "2000" "2008" "1995" "1998" "2014" "1998" "2004" "2013" "2007"n <- 10
selection <- c(letters, "&", "_")
noisystring2 <- paste0(makenoise(n, selection), sample(1990:2015, n, rep=T), makenoise(n, selection))
## ------------------------------------------------------------------------
noisystring2## [1] "c_ed1992flx" "yanf1998bls" "auhk2001dnrz" "rfywo2007yeiab"
## [5] "ovggg2001_hxc" "zeb2008wurg" "xgrh2015fa" "xzdu1991mamu"
## [9] "_ti2013xlb" "tbsz2000_d"## ------------------------------------------------------------------------
gsub("[^0-9]", "", noisystring2)## [1] "1992" "1998" "2001" "2007" "2001" "2008" "2015" "1991" "2013" "2000"noisydata <- data_frame(noisystring,noisystring2)
noisydata$noisydata_numbers <- gsub("[^0-9]", "", noisydata$noisystring2)
noisydata$noisydata_letters <- gsub("[0-9]", "", noisydata$noisystring2)
noisydata## # A tibble: 10 x 4
## noisystring noisystring2 noisydata_numbers noisydata_letters
## <chr> <chr> <chr> <chr>
## 1 rei1996lpe c_ed1992flx 1992 c_edflx
## 2 zxgf2000ijhy yanf1998bls 1998 yanfbls
## 3 knglb2008isxqp auhk2001dnrz 2001 auhkdnrz
## 4 fk1995pbr rfywo2007yeiab 2007 rfywoyeiab
## 5 tgh1998pmpd ovggg2001_hxc 2001 ovggg_hxc
## 6 e2014tebyd zeb2008wurg 2008 zebwurg
## 7 ku1998ifxa xgrh2015fa 2015 xgrhfa
## 8 nboc2004qtngg xzdu1991mamu 1991 xzdumamu
## 9 vzrc2013hpc _ti2013xlb 2013 _tixlb
## 10 kam2007lb tbsz2000_d 2000 tbsz_d1.1.4 Faktorit
Faktoreilla kuvataan kategorisia/järjestysasteikollisia muuttujia, jonka arvoilla on tietty järjestys. Faktoreita voi muodostaa joko funktioilla factor() tai readr::parse_factor().
library(forcats)
x1 <- c("Joulu", "Huhti", "Tammi", "Maalis")
kk_levelit <- c(
"Tammi", "Helmi", "Maalis", "Huhti", "Touko", "Kesä",
"Heinä", "Elo", "Syys", "Loka", "Marras", "Joulu"
)
factor(x1, kk_levelit)## [1] Joulu Huhti Tammi Maalis
## 12 Levels: Tammi Helmi Maalis Huhti Touko Kesä Heinä Elo Syys ... Joulureadr::parse_factor(x1, kk_levelit)## [1] Joulu Huhti Tammi Maalis
## 12 Levels: Tammi Helmi Maalis Huhti Touko Kesä Heinä Elo Syys ... JouluFaktorit ja starwars-data
library(tidyverse)
sw <- dplyr::starwars
# kirjainvektori
ggplot(sw, aes(x=name,y=height)) +
geom_point() +
theme(axis.text = element_text(angle=90))
# faktori
sw$name <- as.factor(sw$name) #
ggplot(sw, aes(x=name,y=height)) +
geom_point() +
theme(axis.text = element_text(angle=90))
# reorder
sw$name <- fct_reorder(f = sw$name, x = sw$height) #
ggplot(sw, aes(x=name,y=height)) +
geom_point() +
theme(axis.text = element_text(angle=90))
# relevel
f <- factor(c("a", "b", "c"))
fct_relevel(f)
fct_relevel(f, "c")
fct_relevel(f, "b", "a")
# reverse
fct_rev(f)1.1.5 Datojen yhdistäminen
Rivien pinoaminen dplyr::bind_rows()
library(tidyverse)
library(rqog)
oecd <- read_qog(which.data = "oecd", data.type = "time-series")
# Valitaa pari muutama muuttuja
oecd_subset <- oecd %>% select(cname,year,contains("imf_"))
dim(oecd_subset)## [1] 2520 20# suomidata
d_fi <- oecd_subset %>% filter(cname == "Finland")
d_fi## cname year imf_ab imf_abd imf_exp imf_expg imf_gd imf_gdp imf_gdpgr
## 1 Finland 1946 NA NA NA NA NA NA NA
## 2 Finland 1947 NA NA NA NA NA NA NA
## 3 Finland 1948 NA NA NA NA NA NA NA
## 4 Finland 1949 NA NA NA NA NA NA NA
## 5 Finland 1950 NA NA NA NA NA NA NA
## 6 Finland 1951 NA NA NA NA NA NA NA
## 7 Finland 1952 NA NA NA NA NA NA NA
## 8 Finland 1953 NA NA NA NA NA NA NA
## 9 Finland 1954 NA NA NA NA NA NA NA
## 10 Finland 1955 NA NA NA NA NA NA NA
## 11 Finland 1956 NA NA NA NA NA NA NA
## 12 Finland 1957 NA NA NA NA NA NA NA
## 13 Finland 1958 NA NA NA NA NA NA NA
## 14 Finland 1959 NA NA NA NA NA NA NA
## 15 Finland 1960 NA NA NA NA NA NA NA
## 16 Finland 1961 NA NA NA NA NA NA NA
## 17 Finland 1962 NA NA NA NA NA NA NA
## 18 Finland 1963 NA NA NA NA NA NA NA
## 19 Finland 1964 NA NA NA NA NA NA NA
## 20 Finland 1965 NA NA NA NA NA NA NA
## 21 Finland 1966 NA NA NA NA NA NA NA
## 22 Finland 1967 NA NA NA NA NA NA NA
## 23 Finland 1968 NA NA NA NA NA NA NA
## 24 Finland 1969 NA NA NA NA NA NA NA
## 25 Finland 1970 NA NA NA NA NA NA NA
## 26 Finland 1971 NA NA NA NA NA NA NA
## 27 Finland 1972 NA NA NA NA NA NA NA
## 28 Finland 1973 NA NA NA NA NA NA NA
## 29 Finland 1974 NA NA NA NA NA NA NA
## 30 Finland 1975 NA NA NA NA NA NA NA
## 31 Finland 1976 NA NA NA NA NA NA NA
## 32 Finland 1977 NA NA NA NA NA NA NA
## 33 Finland 1978 NA NA NA NA NA NA NA
## 34 Finland 1979 NA NA NA NA NA NA NA
## 35 Finland 1980 -2.611 -1.403 43.864 NA 10.893 53.731 5.673
## 36 Finland 1981 -0.909 -0.478 44.542 -5.208 11.544 52.625 1.294
## 37 Finland 1982 -1.738 -0.923 46.429 0.963 13.851 53.123 3.087
## 38 Finland 1983 -2.202 -1.124 48.197 3.164 15.379 51.065 3.122
## 39 Finland 1984 -0.039 -0.021 47.519 -1.548 15.180 53.028 3.213
## 40 Finland 1985 -1.434 -0.806 49.399 6.720 15.837 56.234 3.537
## 41 Finland 1986 -0.941 -0.693 50.038 5.291 16.443 73.660 2.726
## 42 Finland 1987 -1.887 -1.731 50.976 7.790 17.649 91.782 3.559
## 43 Finland 1988 -2.476 -2.694 49.546 10.870 16.535 108.806 5.211
## 44 Finland 1989 -4.862 -5.797 46.801 8.364 14.269 119.237 5.087
## 45 Finland 1990 -4.929 -6.990 47.060 -2.300 13.848 141.806 0.679
## 46 Finland 1991 -5.249 -6.734 55.731 -6.903 21.878 128.279 -5.916
## 47 Finland 1992 -4.520 -5.112 60.765 9.459 39.255 113.104 -3.325
## 48 Finland 1993 -1.261 -1.126 63.558 16.689 54.108 89.323 -0.734
## 49 Finland 1994 1.059 1.099 62.124 12.873 56.118 103.761 3.940
## 50 Finland 1995 4.111 5.524 59.970 8.235 55.147 134.350 4.206
## imf_gdpppps imf_gns imf_imp imf_impg imf_infl imf_inflch imf_inv
## 1 NA NA NA NA NA NA NA
## 2 NA NA NA NA NA NA NA
## 3 NA NA NA NA NA NA NA
## 4 NA NA NA NA NA NA NA
## 5 NA NA NA NA NA NA NA
## 6 NA NA NA NA NA NA NA
## 7 NA NA NA NA NA NA NA
## 8 NA NA NA NA NA NA NA
## 9 NA NA NA NA NA NA NA
## 10 NA NA NA NA NA NA NA
## 11 NA NA NA NA NA NA NA
## 12 NA NA NA NA NA NA NA
## 13 NA NA NA NA NA NA NA
## 14 NA NA NA NA NA NA NA
## 15 NA NA NA NA NA NA NA
## 16 NA NA NA NA NA NA NA
## 17 NA NA NA NA NA NA NA
## 18 NA NA NA NA NA NA NA
## 19 NA NA NA NA NA NA NA
## 20 NA NA NA NA NA NA NA
## 21 NA NA NA NA NA NA NA
## 22 NA NA NA NA NA NA NA
## 23 NA NA NA NA NA NA NA
## 24 NA NA NA NA NA NA NA
## 25 NA NA NA NA NA NA NA
## 26 NA NA NA NA NA NA NA
## 27 NA NA NA NA NA NA NA
## 28 NA NA NA NA NA NA NA
## 29 NA NA NA NA NA NA NA
## 30 NA NA NA NA NA NA NA
## 31 NA NA NA NA NA NA NA
## 32 NA NA NA NA NA NA NA
## 33 NA NA NA NA NA NA NA
## 34 NA NA NA NA NA NA NA
## 35 0.342 28.532 8.552 NA 32.979 11.602 31.224
## 36 0.341 27.584 -4.588 7.817 36.936 12.000 28.377
## 37 0.350 26.973 2.210 -0.319 40.371 9.300 28.654
## 38 0.352 26.315 4.113 3.238 43.763 8.400 28.372
## 39 0.348 27.281 0.918 7.006 46.826 7.000 27.208
## 40 0.347 25.610 6.362 1.586 49.551 5.820 26.926
## 41 0.345 25.118 3.492 3.202 50.988 2.900 26.036
## 42 0.344 24.916 8.790 1.594 53.070 4.082 26.788
## 43 0.346 26.614 10.630 3.452 55.795 5.135 29.090
## 44 0.351 27.135 9.012 1.242 59.462 6.572 31.983
## 45 0.339 25.190 -0.341 1.700 62.409 4.957 30.119
## 46 0.311 18.823 -13.319 -16.908 65.246 4.545 24.073
## 47 0.270 16.532 0.700 0.579 67.369 3.254 21.052
## 48 0.263 17.312 1.357 -1.091 69.610 3.326 18.573
## 49 0.266 20.812 12.813 17.798 70.733 1.614 19.754
## 50 0.267 24.393 8.188 6.837 71.015 0.398 20.282
## imf_nlb imf_pop imf_rev imf_ue
## 1 NA NA NA NA
## 2 NA NA NA NA
## 3 NA NA NA NA
## 4 NA NA NA NA
## 5 NA NA NA NA
## 6 NA NA NA NA
## 7 NA NA NA NA
## 8 NA NA NA NA
## 9 NA NA NA NA
## 10 NA NA NA NA
## 11 NA NA NA NA
## 12 NA NA NA NA
## 13 NA NA NA NA
## 14 NA NA NA NA
## 15 NA NA NA NA
## 16 NA NA NA NA
## 17 NA NA NA NA
## 18 NA NA NA NA
## 19 NA NA NA NA
## 20 NA NA NA NA
## 21 NA NA NA NA
## 22 NA NA NA NA
## 23 NA NA NA NA
## 24 NA NA NA NA
## 25 NA NA NA NA
## 26 NA NA NA NA
## 27 NA NA NA NA
## 28 NA NA NA NA
## 29 NA NA NA NA
## 30 NA NA NA NA
## 31 NA NA NA NA
## 32 NA NA NA NA
## 33 NA NA NA NA
## 34 NA NA NA NA
## 35 2.880 4.771 46.745 5.288
## 36 4.779 4.788 49.321 5.743
## 37 2.401 4.812 48.830 6.076
## 38 0.359 4.842 48.555 6.147
## 39 2.668 4.870 50.186 5.928
## 40 2.553 4.894 51.952 6.049
## 41 3.345 4.911 53.383 6.665
## 42 0.719 4.926 51.694 4.900
## 43 4.872 4.939 54.418 4.208
## 44 7.138 4.954 53.939 3.108
## 45 6.630 4.974 53.690 3.167
## 46 0.301 4.998 56.031 6.608
## 47 -4.877 5.029 55.888 11.708
## 48 -8.079 5.055 55.478 16.342
## 49 -6.398 5.078 55.725 16.583
## 50 -5.970 5.099 54.000 15.400
## [ reached getOption("max.print") -- omitted 22 rows ]# ruotsidata
d_se <- oecd %>% filter(cname == "Sweden")
d_se## ccode cname year ccodealp cname_year ccodealp_year ccodecow ccodewb
## version aid_cpnc aid_cpsc aid_crnc aid_crnio aid_crsc
## aid_crsio al_ethnic al_language al_religion arda_angenpct
## arda_bagenpct arda_bugenpct arda_bumahpct arda_buothpct arda_buthrpct
## arda_changpct arda_chcatpct arda_chgenpct arda_chortpct arda_chothpct
## arda_chprtpct arda_cogenpct arda_higenpct arda_isahmpct arda_isalapct
## arda_isgenpct arda_isibdpct arda_islotpct arda_isnatpct arda_isshipct
## arda_issunpct arda_jagenpct arda_jdcnpct arda_jdgenpct arda_jdorpct
## arda_jdotpct arda_jdrfpct arda_norelpct arda_otgenpct arda_shgenpct
## arda_sigenpct arda_sygenpct arda_tagenpct arda_zogenpct bci_bci
## bci_bcistd bicc_gmi bicc_hw bicc_milexp bicc_milper biu_girel
## biu_offrel biu_offres bl_asy15f bl_asy15m bl_asy15mf bl_asy25f
## bl_asy25m bl_asy25mf bl_asyp15f bl_asyp15m bl_asyp15mf bl_asyp25f
## bl_asyp25m bl_asyp25mf bl_asys15f bl_asys15m bl_asys15mf bl_asys25f
## bl_asys25m bl_asys25mf bl_asyt15f bl_asyt15m bl_asyt15mf bl_asyt25f
## bl_asyt25m bl_asyt25mf bl_lh_15f bl_lh_15m bl_lh_15mf bl_lh_25f
## bl_lh_25m bl_lh_25mf bl_lhc_15f bl_lhc_15m bl_lhc_15mf bl_lhc_25f
## bl_lhc_25m bl_lhc_25mf bl_lp_15f bl_lp_15m bl_lp_15mf bl_lp_25f
## bl_lp_25m bl_lp_25mf bl_lpc_15f bl_lpc_15m bl_lpc_15mf bl_lpc_25f
## bl_lpc_25m bl_lpc_25mf bl_ls_15f bl_ls_15m bl_ls_15mf bl_ls_25f
## bl_ls_25m bl_ls_25mf bl_lsc_15f bl_lsc_15m bl_lsc_15mf bl_lsc_25f
## bl_lsc_25m bl_lsc_25mf bl_lu_15f bl_lu_15m bl_lu_15mf bl_lu_25f
## bl_lu_25m bl_lu_25mf bmr_dem bmr_dembr bmr_demdur bmr_demmis
## bmr_demtran bnr_dem cam_contest cam_inclusive ccp_buildsoc ccp_cc
## ccp_childwrk ccp_civil ccp_democ ccp_equal ccp_freerel ccp_hr
## ccp_infoacc ccp_initiat ccp_market ccp_marriage ccp_samesexm ccp_slave
## ccp_socialsm ccp_strike ccp_syst ccp_systyear ccp_taxes chga_demo
## chga_hinst ciri_assn ciri_disap ciri_dommov ciri_elecsd ciri_empinx_new
## ciri_empinx_old ciri_formov ciri_injud ciri_kill ciri_move_old
## ciri_physint ciri_polpris ciri_relfre_new ciri_relfre_old ciri_speech
## ciri_tort ciri_wecon ciri_wopol ciri_worker ciri_wosoc cspf_effect
## cspf_legit cspf_sfi cspv_civviol cspv_civwar cspv_ethviol cspv_ethwar
## cspv_intviol cspv_intwar diat_ati diat_iti diat_ti dpi_auton dpi_cemo
## dpi_checks dpi_cl dpi_dhondt dpi_dmmo dpi_eage dpi_eipc dpi_erlc
## dpi_exelec dpi_finter dpi_fraud dpi_gf dpi_gpage1 dpi_gpage2
## dpi_gprlc1 dpi_gprlc2 dpi_gps1 dpi_gps2 dpi_gps3 dpi_gpvs1 dpi_gpvs2
## dpi_gpvs3 dpi_gs dpi_gvs dpi_hlio dpi_housesys dpi_legelec dpi_lipc
## dpi_maj dpi_mdmh dpi_mt dpi_nogp dpi_nogps dpi_noop dpi_noops dpi_nos
## dpi_numul dpi_ogpvs dpi_opage1 dpi_opf dpi_oprlc1 dpi_plurality
## dpi_polariz dpi_pr dpi_seats dpi_slop1 dpi_slop2 dpi_slop3 dpi_state
## dpi_system dpi_tf dpi_ulprty dpi_vslop1 dpi_vslop2 dpi_vslop3
## dpi_vsoop dpi_vsul dpi_yct dpi_yio dr_eg dr_ig dr_pg dr_sg
## ef_bul ef_carb ef_crop ef_ef ef_fg ef_for ef_gl
## eu_sctrtotpmin eu_scttotn fe_cultdiv fe_etfra fe_plural ffp_dp ffp_eco
## ffp_ext ffp_fe ffp_fsi ffp_gg ffp_hf ffp_hr ffp_ps ffp_ref ffp_sec
## ffp_sl ffp_ued fh_aor fh_cl fh_ep fh_feb fh_fog fh_ipolity2 fh_pair
## fh_polity2 fh_ppp fh_pr fh_rol fh_status fhp_mcei5 fhp_mclr5 fhp_mcpp5
## fhp_score5 fhp_status5 fi_ftradeint fi_ftradeint_pd fi_index
## fi_index_pd fi_legprop fi_legprop_pd fi_reg fi_reg_pd fi_sm
## fi_sm_pd fi_sog fi_sog_pd gd_ptsa gd_ptss gea_ea1524f gea_ea1524m
## gea_ea2534f gea_ea2534m gea_ea3544f gea_ea3544m gea_ea4554f gea_ea4554m
## gea_ea5564f gea_ea5564m gea_ea65f gea_ea65m gle_cgdpc gle_exp
## gle_gdp gle_imp gle_pop gle_rgdpc gle_trade gol_adm gol_dist
## gol_enep gol_enep1 gol_enepo gol_enpp gol_enpp1 gol_enppo gol_est
## gol_est_spec gol_inst gol_mt gol_nos gol_pr gol_preel gol_upseat
## gol_uptier gsd_cg gsd_fr gsd_ia gsd_rg gtm_parl gtm_pr
## gtm_unit h_alignl1 h_f h_j h_l1 h_l2 h_lflo h_polcon3 h_polcon5
## hf_business hf_efiscore hf_financ hf_govint hf_govt hf_invest hf_labor
## hf_monetary hf_prights hf_taxbur hf_trade ht_colonial ht_partsz
## ht_region ht_regtype ht_regtype1 iaep_ae iaep_arr iaep_basp iaep_bp
## iaep_callo iaep_cc iaep_const iaep_constin iaep_constlam iaep_ebbp
## iaep_eccdt iaep_ecdl iaep_eml iaep_enlc iaep_epmf iaep_es iaep_evp
## iaep_ise iaep_lap iaep_lcre iaep_lego iaep_lrit iaep_lvp iaep_milo
## iaep_nee iaep_nel iaep_npa iaep_nr iaep_osp iaep_pm5p iaep_pnlc
## iaep_pselc iaep_pvelc iaep_rbbp iaep_snlc iaep_ufs icrg_qog
## ictd_nontax ictd_rev ictd_soccon ictd_taxcorp ictd_taxexsc ictd_taxgs
## ictd_taxinc ictd_taxind ictd_taxindirect ictd_taxinsc ictd_taxnresexsc
## ictd_taxnresinsc ictd_taxother ictd_taxpaywf ictd_taxprop ictd_taxres
## ictd_taxtrade ihme_drbs ihme_drf ihme_drm ihme_lebs0001 ihme_lebs0104
## ihme_lebs0509 ihme_lebs1014 ihme_lebs1519 ihme_lebs2024 ihme_lebs2529
## ihme_lebs3034 ihme_lebs3539 ihme_lebs4044 ihme_lebs4549 ihme_lebs5054
## ihme_lebs5559 ihme_lebs6064 ihme_lebs6569 ihme_lebs7074 ihme_lebs7579
## ihme_lebs8084 ihme_lef0001 ihme_lef0104 ihme_lef0509 ihme_lef1014
## ihme_lef1519 ihme_lef2024 ihme_lef2529 ihme_lef3034 ihme_lef3539
## ihme_lef4044 ihme_lef4549 ihme_lef5054 ihme_lef5559 ihme_lef6064
## ihme_lef6569 ihme_lef7074 ihme_lef7579 ihme_lef8084 ihme_lem0001
## ihme_lem0104 ihme_lem0509 ihme_lem1014 ihme_lem1519 ihme_lem2024
## ihme_lem2529 ihme_lem3034 ihme_lem3539 ihme_lem4044 ihme_lem4549
## ihme_lem5054 ihme_lem5559 ihme_lem6064 ihme_lem6569 ihme_lem7074
## ihme_lem7579 ihme_lem8084 imf_ab imf_abd imf_exp imf_expg imf_gd
## imf_gdp imf_gdpgr imf_gdpppps imf_gns imf_imp imf_impg imf_infl
## imf_inflch imf_inv imf_nlb imf_pop imf_rev imf_ue ipu_l_s ipu_l_sw
## ipu_l_w jw_avgballot jw_avgpool jw_avgvote jw_bicameral jw_domr
## jw_election jw_indy jw_legsize jw_mdist jw_multiround jw_multitier
## jw_oneparty jw_persr jw_propcoded jw_propmmd jw_propn jw_propsmd
## jw_rank jw_tiervote kun_cluster kun_ecoabs kun_ecorel kun_legabs
## kun_legrel kun_polabs kun_polrel kun_wiqreco_all kun_wiqrleg_all
## kun_wiqrpol_all mad_gdppc nelda_fme nelda_mbbe nelda_mtop nelda_noe
## nelda_noea nelda_noee nelda_noel nelda_oa nelda_rpae nelda_vcdbe no_ce
## no_ef no_ufs oecd_airqty_t1 oecd_doctor_g1 oecd_doctor_g3
## oecd_evogdp_t1 oecd_evopop_t1 oecd_evova_t1a oecd_evova_t1b
## oecd_evova_t1d oecd_evova_t1e oecd_evova_t1f oecd_evova_t1g
## oecd_housinc_t1 oecd_houssave_t1 oecd_invrates_t1 oecd_migeduemp_t1c
## oecd_migeduemp_t1f oecd_migforpop_t1a oecd_natinccap_t1 oecd_nurse_g1
## oecd_nurse_g3 oecd_prodincom_g1 oecd_prodincom_g2a oecd_prodincom_g2b
## oecd_prodincom_g2c oecd_smoke_g1 oecd_soxnox_t1a oecd_soxnox_t1b
## oecd_valaddac_t1a oecd_valaddac_t1b oecd_valaddac_t1c oecd_valaddac_t1d
## oecd_valaddac_t1e oecd_valaddac_t1f oecd_valaddac_t1g oecd_waste_t1b
## p_autoc p_democ p_durable p_flag p_fragment p_parcomp p_parreg p_polity
## p_polity2 p_xconst p_xrcomp p_xropen p_xrreg pwt_cs pwt_csppp
## pwt_gc pwt_hci pwt_me pwt_mi pwt_plcf pwt_plcs pwt_ple
## pwt_plgc pwt_plhc pwt_pli pwt_pop pwt_rgdp pwt_rt
## pwt_sgcf pwt_shhc pwt_slcgdp pwt_tfp pwt_tfpppp pwt_xr r_elf85
## ross_gas_exp ross_gas_netexp ross_gas_netexpc ross_gas_price
## ross_gas_prod ross_gas_value_2000 ross_gas_value_2014 ross_oil_exp
## ross_oil_netexp ross_oil_netexpc ross_oil_price ross_oil_prod
## ross_oil_value_2000 ross_oil_value_2014 rsf_pfi shec_se ti_cpi
## ti_cpi_max ti_cpi_min uds_mean uds_median uds_pct025 uds_pct975
## uds_sd undp_hdi une_girg1pf une_girg1pm une_girg1pt une_oeals
## une_oeapsnt une_oeaus une_tdurls une_tdurpsnt une_tdurused unna_ahff
## unna_cii unna_con unna_er unna_fce unna_gcf unna_gdp
## unna_gdppc unna_gfcf unna_ggfce unna_gse unna_gsi unna_hce unna_man
## unna_mmu unna_oa unna_pop unna_tsc unna_wrrh vdem_corr vdem_delibdem
## vdem_dl_delib vdem_edcomp_thick vdem_egal vdem_egaldem vdem_elvotbuy
## vdem_exbribe vdem_excrptps vdem_execorr vdem_exembez vdem_exthftps
## vdem_gcrrpt vdem_gender vdem_jucorrdc vdem_libdem vdem_liberal
## vdem_mecorrpt vdem_partip vdem_partipdem vdem_polyarchy vdem_pubcorr
## vi_ext vi_mws vi_nmw vi_rag vi_ram vi_rcbg vi_rcbm vi_rsg vi_rsm
## vi_udr vi_wcoord vi_wgi vi_wl voh_gti wbgi_cce wbgi_ccn wbgi_ccs
## wbgi_gee wbgi_gen wbgi_ges wbgi_pve wbgi_pvn wbgi_pvs wbgi_rle
## wbgi_rln wbgi_rls wbgi_rqe wbgi_rqn wbgi_rqs wbgi_vae wbgi_van
## wbgi_vas wdi_accf wdi_acel wdi_acelr wdi_acelu wdi_acis wdi_acisr
## wdi_acisu wdi_aciw wdi_aciwr wdi_aciwu wdi_afp wdi_afpt wdi_agedr
## wdi_ane wdi_araland wdi_area wdi_armexp wdi_armimp wdi_birth
## wdi_birthskill wdi_broadb wdi_co2 wdi_death wdi_debt wdi_eduprp
## wdi_eduprs wdi_elerenew wdi_elprodcoal wdi_elprodgas wdi_elprodhyd
## wdi_elprodnuc wdi_elprodoil wdi_emp wdi_empagr wdi_empagrf wdi_empagrm
## wdi_empf wdi_empind wdi_empindf wdi_empindm wdi_empm wdi_empprfilo
## wdi_empprfne wdi_empprilo wdi_empprmilo wdi_empprmne wdi_empprne
## wdi_emppryfilo wdi_emppryfne wdi_emppryilo wdi_empprymilo wdi_empprymne
## wdi_emppryne wdi_empser wdi_empserf wdi_empserm wdi_eneimp wdi_enerenew
## wdi_eneuse wdi_expedu wdi_expeduge wdi_expedup wdi_expedus wdi_expedut
## wdi_exph wdi_exphpr wdi_exphpu wdi_exphpuge wdi_exphpuhe wdi_expmil
## wdi_expmilge wdi_export wdi_expstup wdi_expstus wdi_expstut
## wdi_fdiin wdi_fdiout wdi_fertility wdi_forest wdi_fossil wdi_gdpagr
## wdi_gdpcapcon2010 wdi_gdpcapcur wdi_gdpcapgr wdi_gdpcappppcon2011
## wdi_gdpcappppcur wdi_gdpgr wdi_gdpind wdi_gdppppcon2011 wdi_gdppppcur
## wdi_gdpser wdi_gerp wdi_gerpf wdi_gerpm wdi_gerpp wdi_gerppf
## wdi_gerppm wdi_gers wdi_gersf wdi_gersm wdi_gert wdi_gertf wdi_gertm
## wdi_gini wdi_gniatlcur wdi_gnicapatlcur wdi_gnicapcon2010 wdi_gnicapgr
## wdi_gnicappppcon2011 wdi_gnicappppcur wdi_gnicon2010 wdi_gnicur
## wdi_gnigr wdi_gnipppcon2011 wdi_gnipppcur wdi_homicides wdi_import
## wdi_incsh10h wdi_incsh10l wdi_incsh202 wdi_incsh203 wdi_incsh204
## wdi_incsh20h wdi_incsh20l wdi_inflation wdi_interexp wdi_internet
## wdi_interrev wdi_intrate wdi_lfpedua wdi_lfpeduaf wdi_lfpeduam
## wdi_lfpedub wdi_lfpedubf wdi_lfpedubm wdi_lfpedui wdi_lfpeduif
## wdi_lfpeduim wdi_lfpf wdi_lfpfilo15 wdi_lfpfne15 wdi_lfpilo15
## wdi_lfpmilo15 wdi_lfpmne15 wdi_lfpne15 wdi_lfpyfilo wdi_lfpyfne
## wdi_lfpyilo wdi_lfpymilo wdi_lfpymne wdi_lfpyne wdi_lifexp wdi_lifexpf
## wdi_lifexpm wdi_lrmd wdi_migration wdi_mobile wdi_mortf wdi_mortinf
## wdi_mortm wdi_mortnn wdi_mortu5 wdi_nerp wdi_nerpf wdi_nerpm wdi_nerpr
## wdi_nerprf wdi_nerprm wdi_oilrent wdi_pop wdi_pop14 wdi_pop1564
## wdi_pop65 wdi_popden wdi_popf wdi_popgr wdi_poprul wdi_poprulgr
## wdi_popurb wdi_popurbagr wdi_povgap190 wdi_povgap320 wdi_powcon
## wdi_precip wdi_refasy wdi_refori wdi_semp wdi_sempf wdi_sempm
## wdi_taxrev wdi_tele wdi_trade wdi_tradeserv wdi_unempedua
## wdi_unempeduaf wdi_unempeduam wdi_unempedub wdi_unempedubf
## wdi_unempedubm wdi_unempedui wdi_unempeduif wdi_unempeduim
## wdi_unempfilo wdi_unempfne wdi_unempilo wdi_unempmilo wdi_unempmne
## wdi_unempne wdi_unempyfilo wdi_unempyfne wdi_unempyilo wdi_unempymilo
## wdi_unempymne wdi_unempyne wdi_wip wef_aas wef_amp wef_audit wef_bccv
## wef_bct wef_bgr wef_bihiv wef_bit wef_chiv wef_ci wef_ct
## wef_dpf wef_dtsb wef_ebf wef_elec wef_fgo wef_gbb wef_gci wef_gd
## wef_gdpp2 wef_gns wef_ias wef_ilc wef_imort wef_infl wef_ipr
## wef_ji wef_lifexp wef_md wef_mobile wef_oc wef_pr wef_ptp
## wef_ptsb wef_qair wef_qes wef_qoi wef_qpe wef_qport wef_qroad
## wef_qsri wef_rps wef_tax wef_tele wef_tgp wef_uic wef_wgs wef_wlf
## wel_citrig wel_coc wel_dr wel_edi wel_par wel_ppr wel_regtype
## wel_rli wel_rol wel_scalezone wel_sys who_alc1999 who_alc2000
## whr_hap wr_nonautocracy
## [ reached getOption("max.print") -- omitted 72 rows ]# norjadata
d_no <- oecd %>% filter(cname == "Norway")
d_no## ccode cname year ccodealp cname_year ccodealp_year ccodecow ccodewb
## version aid_cpnc aid_cpsc aid_crnc aid_crnio aid_crsc
## aid_crsio al_ethnic al_language al_religion arda_angenpct arda_bagenpct
## arda_bugenpct arda_bumahpct arda_buothpct arda_buthrpct arda_changpct
## arda_chcatpct arda_chgenpct arda_chortpct arda_chothpct arda_chprtpct
## arda_cogenpct arda_higenpct arda_isahmpct arda_isalapct arda_isgenpct
## arda_isibdpct arda_islotpct arda_isnatpct arda_isshipct arda_issunpct
## arda_jagenpct arda_jdcnpct arda_jdgenpct arda_jdorpct arda_jdotpct
## arda_jdrfpct arda_norelpct arda_otgenpct arda_shgenpct arda_sigenpct
## arda_sygenpct arda_tagenpct arda_zogenpct bci_bci bci_bcistd bicc_gmi
## bicc_hw bicc_milexp bicc_milper biu_girel biu_offrel biu_offres
## bl_asy15f bl_asy15m bl_asy15mf bl_asy25f bl_asy25m bl_asy25mf
## bl_asyp15f bl_asyp15m bl_asyp15mf bl_asyp25f bl_asyp25m bl_asyp25mf
## bl_asys15f bl_asys15m bl_asys15mf bl_asys25f bl_asys25m bl_asys25mf
## bl_asyt15f bl_asyt15m bl_asyt15mf bl_asyt25f bl_asyt25m bl_asyt25mf
## bl_lh_15f bl_lh_15m bl_lh_15mf bl_lh_25f bl_lh_25m bl_lh_25mf
## bl_lhc_15f bl_lhc_15m bl_lhc_15mf bl_lhc_25f bl_lhc_25m bl_lhc_25mf
## bl_lp_15f bl_lp_15m bl_lp_15mf bl_lp_25f bl_lp_25m bl_lp_25mf
## bl_lpc_15f bl_lpc_15m bl_lpc_15mf bl_lpc_25f bl_lpc_25m bl_lpc_25mf
## bl_ls_15f bl_ls_15m bl_ls_15mf bl_ls_25f bl_ls_25m bl_ls_25mf
## bl_lsc_15f bl_lsc_15m bl_lsc_15mf bl_lsc_25f bl_lsc_25m bl_lsc_25mf
## bl_lu_15f bl_lu_15m bl_lu_15mf bl_lu_25f bl_lu_25m bl_lu_25mf
## bmr_dem bmr_dembr bmr_demdur bmr_demmis bmr_demtran bnr_dem cam_contest
## cam_inclusive ccp_buildsoc ccp_cc ccp_childwrk ccp_civil ccp_democ
## ccp_equal ccp_freerel ccp_hr ccp_infoacc ccp_initiat ccp_market
## ccp_marriage ccp_samesexm ccp_slave ccp_socialsm ccp_strike ccp_syst
## ccp_systyear ccp_taxes chga_demo chga_hinst ciri_assn ciri_disap
## ciri_dommov ciri_elecsd ciri_empinx_new ciri_empinx_old ciri_formov
## ciri_injud ciri_kill ciri_move_old ciri_physint ciri_polpris
## ciri_relfre_new ciri_relfre_old ciri_speech ciri_tort ciri_wecon
## ciri_wopol ciri_worker ciri_wosoc cspf_effect cspf_legit cspf_sfi
## cspv_civviol cspv_civwar cspv_ethviol cspv_ethwar cspv_intviol
## cspv_intwar diat_ati diat_iti diat_ti dpi_auton dpi_cemo dpi_checks
## dpi_cl dpi_dhondt dpi_dmmo dpi_eage dpi_eipc dpi_erlc dpi_exelec
## dpi_finter dpi_fraud dpi_gf dpi_gpage1 dpi_gpage2 dpi_gprlc1
## dpi_gprlc2 dpi_gps1 dpi_gps2 dpi_gps3 dpi_gpvs1 dpi_gpvs2 dpi_gpvs3
## dpi_gs dpi_gvs dpi_hlio dpi_housesys dpi_legelec dpi_lipc dpi_maj
## dpi_mdmh dpi_mt dpi_nogp dpi_nogps dpi_noop dpi_noops dpi_nos dpi_numul
## dpi_ogpvs dpi_opage1 dpi_opf dpi_oprlc1 dpi_plurality dpi_polariz
## dpi_pr dpi_seats dpi_slop1 dpi_slop2 dpi_slop3 dpi_state dpi_system
## dpi_tf dpi_ulprty dpi_vslop1 dpi_vslop2 dpi_vslop3 dpi_vsoop
## dpi_vsul dpi_yct dpi_yio dr_eg dr_ig dr_pg dr_sg ef_bul
## ef_carb ef_crop ef_ef ef_fg ef_for ef_gl
## eu_sctrtotpmin eu_scttotn fe_cultdiv fe_etfra fe_plural ffp_dp
## ffp_eco ffp_ext ffp_fe ffp_fsi ffp_gg ffp_hf ffp_hr ffp_ps ffp_ref
## ffp_sec ffp_sl ffp_ued fh_aor fh_cl fh_ep fh_feb fh_fog fh_ipolity2
## fh_pair fh_polity2 fh_ppp fh_pr fh_rol fh_status fhp_mcei5 fhp_mclr5
## fhp_mcpp5 fhp_score5 fhp_status5 fi_ftradeint fi_ftradeint_pd fi_index
## fi_index_pd fi_legprop fi_legprop_pd fi_reg fi_reg_pd fi_sm
## fi_sm_pd fi_sog fi_sog_pd gd_ptsa gd_ptss gea_ea1524f gea_ea1524m
## gea_ea2534f gea_ea2534m gea_ea3544f gea_ea3544m gea_ea4554f gea_ea4554m
## gea_ea5564f gea_ea5564m gea_ea65f gea_ea65m gle_cgdpc gle_exp
## gle_gdp gle_imp gle_pop gle_rgdpc gle_trade gol_adm gol_dist
## gol_enep gol_enep1 gol_enepo gol_enpp gol_enpp1 gol_enppo gol_est
## gol_est_spec gol_inst gol_mt gol_nos gol_pr gol_preel gol_upseat
## gol_uptier gsd_cg gsd_fr gsd_ia gsd_rg gtm_parl gtm_pr
## gtm_unit h_alignl1 h_f h_j h_l1 h_l2 h_lflo h_polcon3 h_polcon5
## hf_business hf_efiscore hf_financ hf_govint hf_govt hf_invest hf_labor
## hf_monetary hf_prights hf_taxbur hf_trade ht_colonial ht_partsz
## ht_region ht_regtype ht_regtype1 iaep_ae iaep_arr iaep_basp iaep_bp
## iaep_callo iaep_cc iaep_const iaep_constin iaep_constlam iaep_ebbp
## iaep_eccdt iaep_ecdl iaep_eml iaep_enlc iaep_epmf iaep_es iaep_evp
## iaep_ise iaep_lap iaep_lcre iaep_lego iaep_lrit iaep_lvp iaep_milo
## iaep_nee iaep_nel iaep_npa iaep_nr iaep_osp iaep_pm5p iaep_pnlc
## iaep_pselc iaep_pvelc iaep_rbbp iaep_snlc iaep_ufs icrg_qog
## ictd_nontax ictd_rev ictd_soccon ictd_taxcorp ictd_taxexsc ictd_taxgs
## ictd_taxinc ictd_taxind ictd_taxindirect ictd_taxinsc ictd_taxnresexsc
## ictd_taxnresinsc ictd_taxother ictd_taxpaywf ictd_taxprop ictd_taxres
## ictd_taxtrade ihme_drbs ihme_drf ihme_drm ihme_lebs0001 ihme_lebs0104
## ihme_lebs0509 ihme_lebs1014 ihme_lebs1519 ihme_lebs2024 ihme_lebs2529
## ihme_lebs3034 ihme_lebs3539 ihme_lebs4044 ihme_lebs4549 ihme_lebs5054
## ihme_lebs5559 ihme_lebs6064 ihme_lebs6569 ihme_lebs7074 ihme_lebs7579
## ihme_lebs8084 ihme_lef0001 ihme_lef0104 ihme_lef0509 ihme_lef1014
## ihme_lef1519 ihme_lef2024 ihme_lef2529 ihme_lef3034 ihme_lef3539
## ihme_lef4044 ihme_lef4549 ihme_lef5054 ihme_lef5559 ihme_lef6064
## ihme_lef6569 ihme_lef7074 ihme_lef7579 ihme_lef8084 ihme_lem0001
## ihme_lem0104 ihme_lem0509 ihme_lem1014 ihme_lem1519 ihme_lem2024
## ihme_lem2529 ihme_lem3034 ihme_lem3539 ihme_lem4044 ihme_lem4549
## ihme_lem5054 ihme_lem5559 ihme_lem6064 ihme_lem6569 ihme_lem7074
## ihme_lem7579 ihme_lem8084 imf_ab imf_abd imf_exp imf_expg imf_gd
## imf_gdp imf_gdpgr imf_gdpppps imf_gns imf_imp imf_impg imf_infl
## imf_inflch imf_inv imf_nlb imf_pop imf_rev imf_ue ipu_l_s ipu_l_sw
## ipu_l_w jw_avgballot jw_avgpool jw_avgvote jw_bicameral jw_domr
## jw_election jw_indy jw_legsize jw_mdist jw_multiround jw_multitier
## jw_oneparty jw_persr jw_propcoded jw_propmmd jw_propn jw_propsmd
## jw_rank jw_tiervote kun_cluster kun_ecoabs kun_ecorel kun_legabs
## kun_legrel kun_polabs kun_polrel kun_wiqreco_all kun_wiqrleg_all
## kun_wiqrpol_all mad_gdppc nelda_fme nelda_mbbe nelda_mtop nelda_noe
## nelda_noea nelda_noee nelda_noel nelda_oa nelda_rpae nelda_vcdbe no_ce
## no_ef no_ufs oecd_airqty_t1 oecd_doctor_g1 oecd_doctor_g3
## oecd_evogdp_t1 oecd_evopop_t1 oecd_evova_t1a oecd_evova_t1b
## oecd_evova_t1d oecd_evova_t1e oecd_evova_t1f oecd_evova_t1g
## oecd_housinc_t1 oecd_houssave_t1 oecd_invrates_t1 oecd_migeduemp_t1c
## oecd_migeduemp_t1f oecd_migforpop_t1a oecd_natinccap_t1 oecd_nurse_g1
## oecd_nurse_g3 oecd_prodincom_g1 oecd_prodincom_g2a oecd_prodincom_g2b
## oecd_prodincom_g2c oecd_smoke_g1 oecd_soxnox_t1a oecd_soxnox_t1b
## oecd_valaddac_t1a oecd_valaddac_t1b oecd_valaddac_t1c oecd_valaddac_t1d
## oecd_valaddac_t1e oecd_valaddac_t1f oecd_valaddac_t1g oecd_waste_t1b
## p_autoc p_democ p_durable p_flag p_fragment p_parcomp p_parreg p_polity
## p_polity2 p_xconst p_xrcomp p_xropen p_xrreg pwt_cs pwt_csppp
## pwt_gc pwt_hci pwt_me pwt_mi pwt_plcf pwt_plcs pwt_ple
## pwt_plgc pwt_plhc pwt_pli pwt_pop pwt_rgdp pwt_rt
## pwt_sgcf pwt_shhc pwt_slcgdp pwt_tfp pwt_tfpppp pwt_xr r_elf85
## ross_gas_exp ross_gas_netexp ross_gas_netexpc ross_gas_price
## ross_gas_prod ross_gas_value_2000 ross_gas_value_2014 ross_oil_exp
## ross_oil_netexp ross_oil_netexpc ross_oil_price ross_oil_prod
## ross_oil_value_2000 ross_oil_value_2014 rsf_pfi shec_se ti_cpi
## ti_cpi_max ti_cpi_min uds_mean uds_median uds_pct025 uds_pct975
## uds_sd undp_hdi une_girg1pf une_girg1pm une_girg1pt une_oeals
## une_oeapsnt une_oeaus une_tdurls une_tdurpsnt une_tdurused unna_ahff
## unna_cii unna_con unna_er unna_fce unna_gcf unna_gdp
## unna_gdppc unna_gfcf unna_ggfce unna_gse unna_gsi unna_hce unna_man
## unna_mmu unna_oa unna_pop unna_tsc unna_wrrh vdem_corr vdem_delibdem
## vdem_dl_delib vdem_edcomp_thick vdem_egal vdem_egaldem vdem_elvotbuy
## vdem_exbribe vdem_excrptps vdem_execorr vdem_exembez vdem_exthftps
## vdem_gcrrpt vdem_gender vdem_jucorrdc vdem_libdem vdem_liberal
## vdem_mecorrpt vdem_partip vdem_partipdem vdem_polyarchy vdem_pubcorr
## vi_ext vi_mws vi_nmw vi_rag vi_ram vi_rcbg vi_rcbm vi_rsg vi_rsm
## vi_udr vi_wcoord vi_wgi vi_wl voh_gti wbgi_cce wbgi_ccn wbgi_ccs
## wbgi_gee wbgi_gen wbgi_ges wbgi_pve wbgi_pvn wbgi_pvs wbgi_rle
## wbgi_rln wbgi_rls wbgi_rqe wbgi_rqn wbgi_rqs wbgi_vae wbgi_van
## wbgi_vas wdi_accf wdi_acel wdi_acelr wdi_acelu wdi_acis wdi_acisr
## wdi_acisu wdi_aciw wdi_aciwr wdi_aciwu wdi_afp wdi_afpt wdi_agedr
## wdi_ane wdi_araland wdi_area wdi_armexp wdi_armimp wdi_birth
## wdi_birthskill wdi_broadb wdi_co2 wdi_death wdi_debt wdi_eduprp
## wdi_eduprs wdi_elerenew wdi_elprodcoal wdi_elprodgas wdi_elprodhyd
## wdi_elprodnuc wdi_elprodoil wdi_emp wdi_empagr wdi_empagrf wdi_empagrm
## wdi_empf wdi_empind wdi_empindf wdi_empindm wdi_empm wdi_empprfilo
## wdi_empprfne wdi_empprilo wdi_empprmilo wdi_empprmne wdi_empprne
## wdi_emppryfilo wdi_emppryfne wdi_emppryilo wdi_empprymilo wdi_empprymne
## wdi_emppryne wdi_empser wdi_empserf wdi_empserm wdi_eneimp wdi_enerenew
## wdi_eneuse wdi_expedu wdi_expeduge wdi_expedup wdi_expedus wdi_expedut
## wdi_exph wdi_exphpr wdi_exphpu wdi_exphpuge wdi_exphpuhe wdi_expmil
## wdi_expmilge wdi_export wdi_expstup wdi_expstus wdi_expstut wdi_fdiin
## wdi_fdiout wdi_fertility wdi_forest wdi_fossil wdi_gdpagr
## wdi_gdpcapcon2010 wdi_gdpcapcur wdi_gdpcapgr wdi_gdpcappppcon2011
## wdi_gdpcappppcur wdi_gdpgr wdi_gdpind wdi_gdppppcon2011 wdi_gdppppcur
## wdi_gdpser wdi_gerp wdi_gerpf wdi_gerpm wdi_gerpp wdi_gerppf
## wdi_gerppm wdi_gers wdi_gersf wdi_gersm wdi_gert wdi_gertf wdi_gertm
## wdi_gini wdi_gniatlcur wdi_gnicapatlcur wdi_gnicapcon2010 wdi_gnicapgr
## wdi_gnicappppcon2011 wdi_gnicappppcur wdi_gnicon2010 wdi_gnicur
## wdi_gnigr wdi_gnipppcon2011 wdi_gnipppcur wdi_homicides wdi_import
## wdi_incsh10h wdi_incsh10l wdi_incsh202 wdi_incsh203 wdi_incsh204
## wdi_incsh20h wdi_incsh20l wdi_inflation wdi_interexp wdi_internet
## wdi_interrev wdi_intrate wdi_lfpedua wdi_lfpeduaf wdi_lfpeduam
## wdi_lfpedub wdi_lfpedubf wdi_lfpedubm wdi_lfpedui wdi_lfpeduif
## wdi_lfpeduim wdi_lfpf wdi_lfpfilo15 wdi_lfpfne15 wdi_lfpilo15
## wdi_lfpmilo15 wdi_lfpmne15 wdi_lfpne15 wdi_lfpyfilo wdi_lfpyfne
## wdi_lfpyilo wdi_lfpymilo wdi_lfpymne wdi_lfpyne wdi_lifexp wdi_lifexpf
## wdi_lifexpm wdi_lrmd wdi_migration wdi_mobile wdi_mortf
## wdi_mortinf wdi_mortm wdi_mortnn wdi_mortu5 wdi_nerp wdi_nerpf
## wdi_nerpm wdi_nerpr wdi_nerprf wdi_nerprm wdi_oilrent wdi_pop
## wdi_pop14 wdi_pop1564 wdi_pop65 wdi_popden wdi_popf wdi_popgr
## wdi_poprul wdi_poprulgr wdi_popurb wdi_popurbagr wdi_povgap190
## wdi_povgap320 wdi_powcon wdi_precip wdi_refasy wdi_refori wdi_semp
## wdi_sempf wdi_sempm wdi_taxrev wdi_tele wdi_trade wdi_tradeserv
## wdi_unempedua wdi_unempeduaf wdi_unempeduam wdi_unempedub
## wdi_unempedubf wdi_unempedubm wdi_unempedui wdi_unempeduif
## wdi_unempeduim wdi_unempfilo wdi_unempfne wdi_unempilo wdi_unempmilo
## wdi_unempmne wdi_unempne wdi_unempyfilo wdi_unempyfne wdi_unempyilo
## wdi_unempymilo wdi_unempymne wdi_unempyne wdi_wip wef_aas wef_amp
## wef_audit wef_bccv wef_bct wef_bgr wef_bihiv wef_bit wef_chiv
## wef_ci wef_ct wef_dpf wef_dtsb wef_ebf wef_elec wef_fgo wef_gbb
## wef_gci wef_gd wef_gdpp2 wef_gns wef_ias wef_ilc wef_imort
## wef_infl wef_ipr wef_ji wef_lifexp wef_md wef_mobile wef_oc
## wef_pr wef_ptp wef_ptsb wef_qair wef_qes wef_qoi wef_qpe
## wef_qport wef_qroad wef_qsri wef_rps wef_tax wef_tele wef_tgp
## wef_uic wef_wgs wef_wlf wel_citrig wel_coc wel_dr wel_edi
## wel_par wel_ppr wel_regtype wel_rli wel_rol wel_scalezone wel_sys
## who_alc1999 who_alc2000 whr_hap wr_nonautocracy
## [ reached getOption("max.print") -- omitted 72 rows ]Pinoa kolme dataa
d_skand <- bind_rows(d_fi, d_se, d_no)Sarakkeiden liittäminen rinnakkain dplyr::bind_cols()
oecd <- read_qog(which.data = "oecd", data.type = "time-series")
# Valitaa pari muutama muuttuja
fi_wdi <- oecd %>% select(cname,year,contains("wdi_")) %>% filter(cname == "Finland")
fi_imf <- oecd %>% select(cname,year,contains("imf_")) %>% filter(cname == "Finland")
bind_cols(fi_wdi,fi_imf)## cname year wdi_accf wdi_acel wdi_acelr wdi_acelu wdi_acis wdi_acisr
## 1 Finland 1946 NA NA NA NA NA NA
## 2 Finland 1947 NA NA NA NA NA NA
## 3 Finland 1948 NA NA NA NA NA NA
## 4 Finland 1949 NA NA NA NA NA NA
## wdi_acisu wdi_aciw wdi_aciwr wdi_aciwu wdi_afp wdi_afpt wdi_agedr
## 1 NA NA NA NA NA NA NA
## 2 NA NA NA NA NA NA NA
## 3 NA NA NA NA NA NA NA
## 4 NA NA NA NA NA NA NA
## wdi_ane wdi_araland wdi_area wdi_armexp wdi_armimp wdi_birth
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_birthskill wdi_broadb wdi_co2 wdi_death wdi_debt wdi_eduprp
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_eduprs wdi_elerenew wdi_elprodcoal wdi_elprodgas wdi_elprodhyd
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_elprodnuc wdi_elprodoil wdi_emp wdi_empagr wdi_empagrf wdi_empagrm
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_empf wdi_empind wdi_empindf wdi_empindm wdi_empm wdi_empprfilo
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_empprfne wdi_empprilo wdi_empprmilo wdi_empprmne wdi_empprne
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_emppryfilo wdi_emppryfne wdi_emppryilo wdi_empprymilo wdi_empprymne
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_emppryne wdi_empser wdi_empserf wdi_empserm wdi_eneimp wdi_enerenew
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_eneuse wdi_expedu wdi_expeduge wdi_expedup wdi_expedus wdi_expedut
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_exph wdi_exphpr wdi_exphpu wdi_exphpuge wdi_exphpuhe wdi_expmil
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_expmilge wdi_export wdi_expstup wdi_expstus wdi_expstut
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_fdiin wdi_fdiout wdi_fertility wdi_forest wdi_fossil wdi_gdpagr
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_gdpcapcon2010 wdi_gdpcapcur wdi_gdpcapgr wdi_gdpcappppcon2011
## 1 NA NA NA NA
## 2 NA NA NA NA
## 3 NA NA NA NA
## 4 NA NA NA NA
## wdi_gdpcappppcur wdi_gdpgr wdi_gdpind wdi_gdppppcon2011 wdi_gdppppcur
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_gdpser wdi_gerp wdi_gerpf wdi_gerpm wdi_gerpp wdi_gerppf
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_gerppm wdi_gers wdi_gersf wdi_gersm wdi_gert wdi_gertf wdi_gertm
## 1 NA NA NA NA NA NA NA
## 2 NA NA NA NA NA NA NA
## 3 NA NA NA NA NA NA NA
## 4 NA NA NA NA NA NA NA
## wdi_gini wdi_gniatlcur wdi_gnicapatlcur wdi_gnicapcon2010 wdi_gnicapgr
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_gnicappppcon2011 wdi_gnicappppcur wdi_gnicon2010 wdi_gnicur
## 1 NA NA NA NA
## 2 NA NA NA NA
## 3 NA NA NA NA
## 4 NA NA NA NA
## wdi_gnigr wdi_gnipppcon2011 wdi_gnipppcur wdi_homicides wdi_import
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_incsh10h wdi_incsh10l wdi_incsh202 wdi_incsh203 wdi_incsh204
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_incsh20h wdi_incsh20l wdi_inflation wdi_interexp wdi_internet
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_interrev wdi_intrate wdi_lfpedua wdi_lfpeduaf wdi_lfpeduam
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_lfpedub wdi_lfpedubf wdi_lfpedubm wdi_lfpedui wdi_lfpeduif
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_lfpeduim wdi_lfpf wdi_lfpfilo15 wdi_lfpfne15 wdi_lfpilo15
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_lfpmilo15 wdi_lfpmne15 wdi_lfpne15 wdi_lfpyfilo wdi_lfpyfne
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_lfpyilo wdi_lfpymilo wdi_lfpymne wdi_lfpyne wdi_lifexp wdi_lifexpf
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_lifexpm wdi_lrmd wdi_migration wdi_mobile wdi_mortf wdi_mortinf
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_mortm wdi_mortnn wdi_mortu5 wdi_nerp wdi_nerpf wdi_nerpm wdi_nerpr
## 1 NA NA NA NA NA NA NA
## 2 NA NA NA NA NA NA NA
## 3 NA NA NA NA NA NA NA
## 4 NA NA NA NA NA NA NA
## wdi_nerprf wdi_nerprm wdi_oilrent wdi_pop wdi_pop14 wdi_pop1564
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_pop65 wdi_popden wdi_popf wdi_popgr wdi_poprul wdi_poprulgr
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_popurb wdi_popurbagr wdi_povgap190 wdi_povgap320 wdi_powcon
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_precip wdi_refasy wdi_refori wdi_semp wdi_sempf wdi_sempm
## 1 NA NA NA NA NA NA
## 2 NA NA NA NA NA NA
## 3 NA NA NA NA NA NA
## 4 NA NA NA NA NA NA
## wdi_taxrev wdi_tele wdi_trade wdi_tradeserv wdi_unempedua
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_unempeduaf wdi_unempeduam wdi_unempedub wdi_unempedubf
## 1 NA NA NA NA
## 2 NA NA NA NA
## 3 NA NA NA NA
## 4 NA NA NA NA
## wdi_unempedubm wdi_unempedui wdi_unempeduif wdi_unempeduim
## 1 NA NA NA NA
## 2 NA NA NA NA
## 3 NA NA NA NA
## 4 NA NA NA NA
## wdi_unempfilo wdi_unempfne wdi_unempilo wdi_unempmilo wdi_unempmne
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_unempne wdi_unempyfilo wdi_unempyfne wdi_unempyilo wdi_unempymilo
## 1 NA NA NA NA NA
## 2 NA NA NA NA NA
## 3 NA NA NA NA NA
## 4 NA NA NA NA NA
## wdi_unempymne wdi_unempyne wdi_wip cname1 year1 imf_ab imf_abd imf_exp
## 1 NA NA NA Finland 1946 NA NA NA
## 2 NA NA NA Finland 1947 NA NA NA
## 3 NA NA NA Finland 1948 NA NA NA
## 4 NA NA NA Finland 1949 NA NA NA
## imf_expg imf_gd imf_gdp imf_gdpgr imf_gdpppps imf_gns imf_imp imf_impg
## 1 NA NA NA NA NA NA NA NA
## 2 NA NA NA NA NA NA NA NA
## 3 NA NA NA NA NA NA NA NA
## 4 NA NA NA NA NA NA NA NA
## imf_infl imf_inflch imf_inv imf_nlb imf_pop imf_rev imf_ue
## 1 NA NA NA NA NA NA NA
## 2 NA NA NA NA NA NA NA
## 3 NA NA NA NA NA NA NA
## 4 NA NA NA NA NA NA NA
## [ reached getOption("max.print") -- omitted 68 rows ]Datojen yhdistäminen avainmuuttujilla dplyr::join
library(dplyr)
filmit <- dplyr::starwars %>% select(name,films) %>% unnest() %>% filter(grepl("^The", films))
filmit## # A tibble: 61 x 2
## name films
## <chr> <chr>
## 1 Luke Skywalker The Empire Strikes Back
## 2 Luke Skywalker The Force Awakens
## 3 C-3PO The Phantom Menace
## 4 C-3PO The Empire Strikes Back
## 5 R2-D2 The Phantom Menace
## 6 R2-D2 The Empire Strikes Back
## 7 R2-D2 The Force Awakens
## 8 Darth Vader The Empire Strikes Back
## 9 Leia Organa The Empire Strikes Back
## 10 Leia Organa The Force Awakens
## # ... with 51 more rowsajoneuvot <- dplyr::starwars %>% select(name,vehicles) %>% unnest()
ajoneuvot## # A tibble: 13 x 2
## name vehicles
## <chr> <chr>
## 1 Luke Skywalker Snowspeeder
## 2 Luke Skywalker Imperial Speeder Bike
## 3 Leia Organa Imperial Speeder Bike
## 4 Obi-Wan Kenobi Tribubble bongo
## 5 Anakin Skywalker Zephyr-G swoop bike
## 6 Anakin Skywalker XJ-6 airspeeder
## 7 Chewbacca AT-ST
## 8 Wedge Antilles Snowspeeder
## 9 Qui-Gon Jinn Tribubble bongo
## 10 Darth Maul Sith speeder
## 11 Dooku Flitknot speeder
## 12 Zam Wesell Koro-2 Exodrive airspeeder
## 13 Grievous Tsmeu-6 personal wheel bikeihmiset <- dplyr::starwars %>% select(name,height,mass,species) %>% filter(species == "Human")
ihmiset## # A tibble: 35 x 4
## name height mass species
## <chr> <int> <dbl> <chr>
## 1 Luke Skywalker 172 77. Human
## 2 Darth Vader 202 136. Human
## 3 Leia Organa 150 49. Human
## 4 Owen Lars 178 120. Human
## 5 Beru Whitesun lars 165 75. Human
## 6 Biggs Darklighter 183 84. Human
## 7 Obi-Wan Kenobi 182 77. Human
## 8 Anakin Skywalker 188 84. Human
## 9 Wilhuff Tarkin 180 NA Human
## 10 Han Solo 180 80. Human
## # ... with 25 more rowsleft_join(ihmiset, filmit, by = "name")## # A tibble: 40 x 5
## name height mass species films
## <chr> <int> <dbl> <chr> <chr>
## 1 Luke Skywalker 172 77. Human The Empire Strikes Back
## 2 Luke Skywalker 172 77. Human The Force Awakens
## 3 Darth Vader 202 136. Human The Empire Strikes Back
## 4 Leia Organa 150 49. Human The Empire Strikes Back
## 5 Leia Organa 150 49. Human The Force Awakens
## 6 Owen Lars 178 120. Human <NA>
## 7 Beru Whitesun lars 165 75. Human <NA>
## 8 Biggs Darklighter 183 84. Human <NA>
## 9 Obi-Wan Kenobi 182 77. Human The Phantom Menace
## 10 Obi-Wan Kenobi 182 77. Human The Empire Strikes Back
## # ... with 30 more rowsright_join(ihmiset, filmit, by = "name")## # A tibble: 61 x 5
## name height mass species films
## <chr> <int> <dbl> <chr> <chr>
## 1 Luke Skywalker 172 77. Human The Empire Strikes Back
## 2 Luke Skywalker 172 77. Human The Force Awakens
## 3 C-3PO NA NA <NA> The Phantom Menace
## 4 C-3PO NA NA <NA> The Empire Strikes Back
## 5 R2-D2 NA NA <NA> The Phantom Menace
## 6 R2-D2 NA NA <NA> The Empire Strikes Back
## 7 R2-D2 NA NA <NA> The Force Awakens
## 8 Darth Vader 202 136. Human The Empire Strikes Back
## 9 Leia Organa 150 49. Human The Empire Strikes Back
## 10 Leia Organa 150 49. Human The Force Awakens
## # ... with 51 more rowsinner_join(ihmiset, filmit, by = "name")## # A tibble: 24 x 5
## name height mass species films
## <chr> <int> <dbl> <chr> <chr>
## 1 Luke Skywalker 172 77. Human The Empire Strikes Back
## 2 Luke Skywalker 172 77. Human The Force Awakens
## 3 Darth Vader 202 136. Human The Empire Strikes Back
## 4 Leia Organa 150 49. Human The Empire Strikes Back
## 5 Leia Organa 150 49. Human The Force Awakens
## 6 Obi-Wan Kenobi 182 77. Human The Phantom Menace
## 7 Obi-Wan Kenobi 182 77. Human The Empire Strikes Back
## 8 Anakin Skywalker 188 84. Human The Phantom Menace
## 9 Han Solo 180 80. Human The Empire Strikes Back
## 10 Han Solo 180 80. Human The Force Awakens
## # ... with 14 more rowsfull_join(ihmiset, filmit, by = "name")## # A tibble: 77 x 5
## name height mass species films
## <chr> <int> <dbl> <chr> <chr>
## 1 Luke Skywalker 172 77. Human The Empire Strikes Back
## 2 Luke Skywalker 172 77. Human The Force Awakens
## 3 Darth Vader 202 136. Human The Empire Strikes Back
## 4 Leia Organa 150 49. Human The Empire Strikes Back
## 5 Leia Organa 150 49. Human The Force Awakens
## 6 Owen Lars 178 120. Human <NA>
## 7 Beru Whitesun lars 165 75. Human <NA>
## 8 Biggs Darklighter 183 84. Human <NA>
## 9 Obi-Wan Kenobi 182 77. Human The Phantom Menace
## 10 Obi-Wan Kenobi 182 77. Human The Empire Strikes Back
## # ... with 67 more rowssemi_join(filmit, ihmiset, by = "name")## # A tibble: 24 x 2
## name films
## <chr> <chr>
## 1 Luke Skywalker The Empire Strikes Back
## 2 Luke Skywalker The Force Awakens
## 3 Darth Vader The Empire Strikes Back
## 4 Leia Organa The Empire Strikes Back
## 5 Leia Organa The Force Awakens
## 6 Obi-Wan Kenobi The Phantom Menace
## 7 Obi-Wan Kenobi The Empire Strikes Back
## 8 Anakin Skywalker The Phantom Menace
## 9 Han Solo The Empire Strikes Back
## 10 Han Solo The Force Awakens
## # ... with 14 more rowsanti_join(filmit, ihmiset, by = "name")## # A tibble: 37 x 2
## name films
## <chr> <chr>
## 1 C-3PO The Phantom Menace
## 2 C-3PO The Empire Strikes Back
## 3 R2-D2 The Phantom Menace
## 4 R2-D2 The Empire Strikes Back
## 5 R2-D2 The Force Awakens
## 6 Chewbacca The Empire Strikes Back
## 7 Chewbacca The Force Awakens
## 8 Jabba Desilijic Tiure The Phantom Menace
## 9 Yoda The Phantom Menace
## 10 Yoda The Empire Strikes Back
## # ... with 27 more rowsLisäverbejä joineihin
dplyr::bind_cols(x,y)
dplyr::bind_rows
dplyr::union
dplyr::intersect
dplyr::setdif1.2 Visualisoinnit
geoms
frekvenssit
library(tidyverse)
sw <- dplyr::starwars
ggplot(sw, aes(species)) + geom_bar()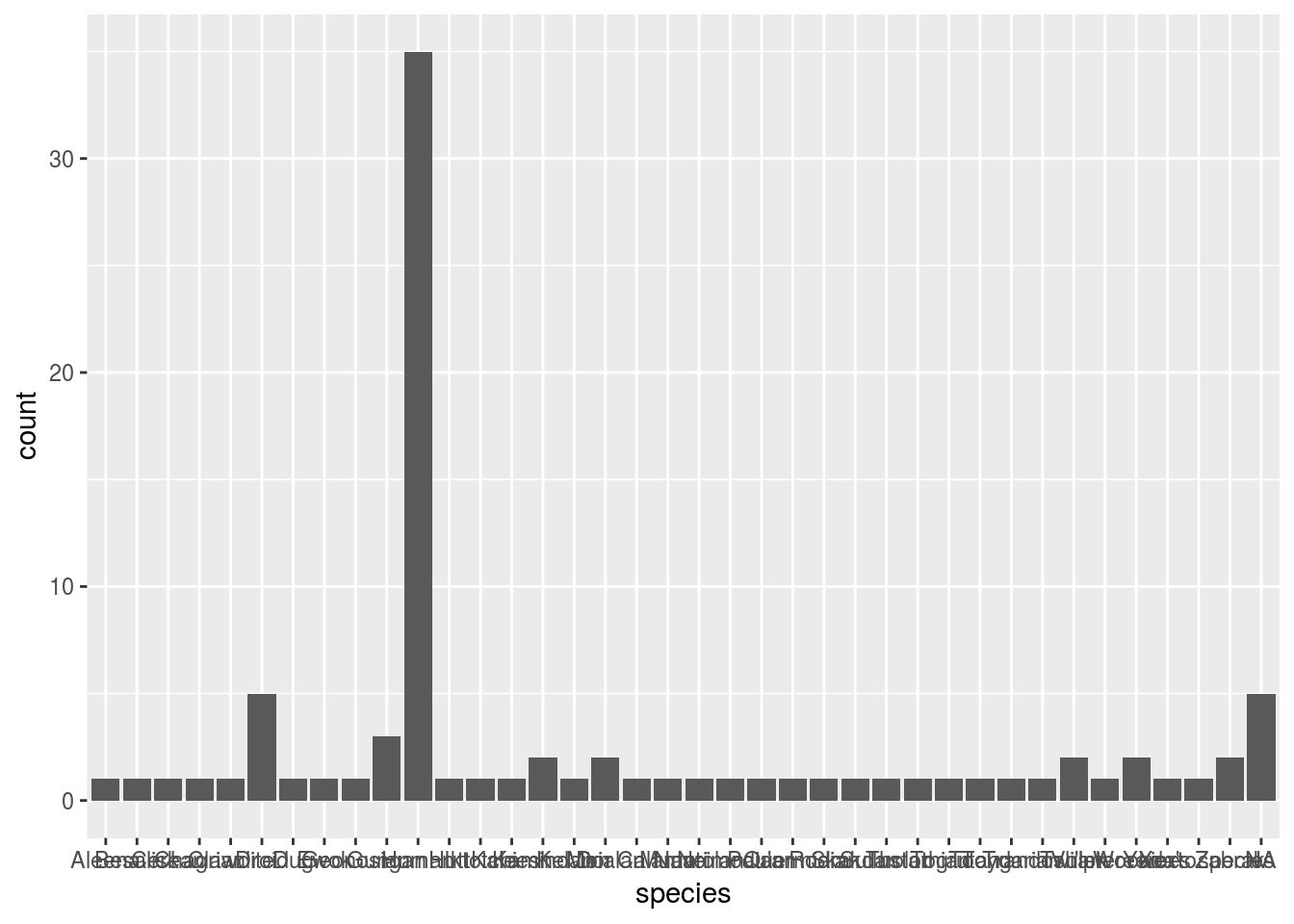
sw$species <- fct_infreq(sw$species)
ggplot(sw, aes(species)) + geom_bar()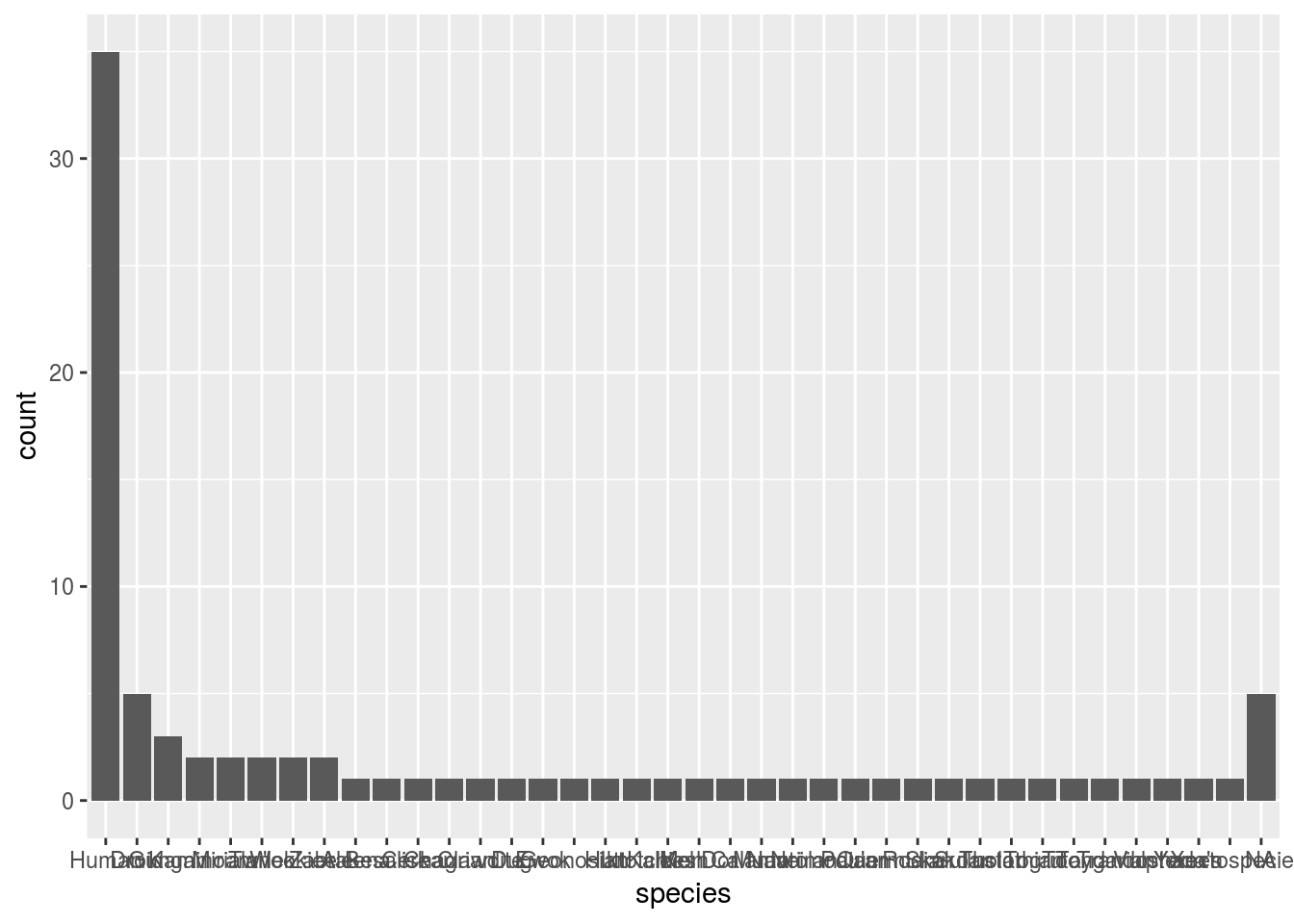
sw$species <- as.character(sw$species)
sw2 <- sw %>% count(species)
ggplot(sw2, aes(x = species, y = n)) + geom_bar(stat = "identity")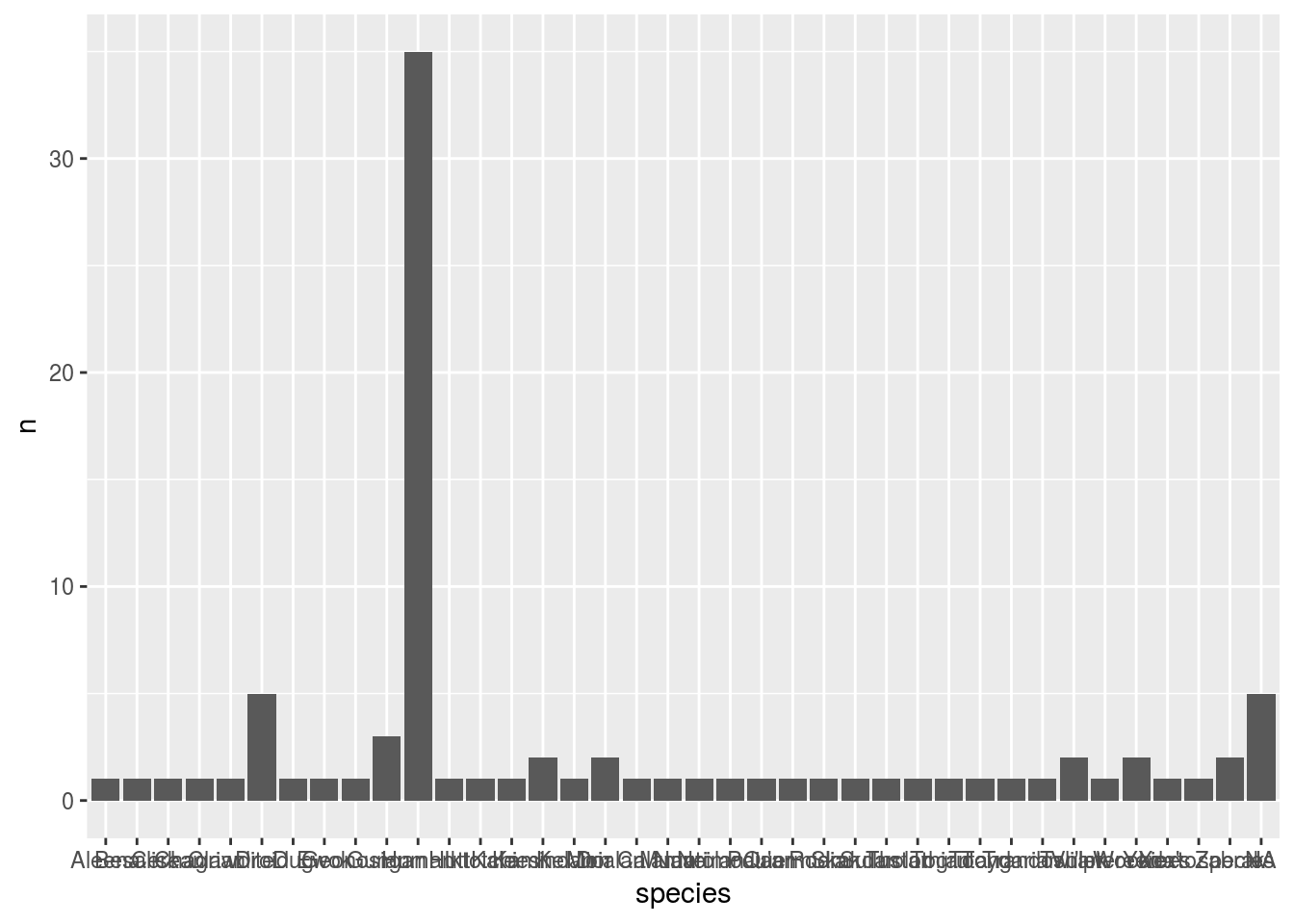
sw2 <- sw %>% count(species)
ggplot(sw2, aes(x = reorder(species, n), y = n)) + geom_bar(stat = "identity")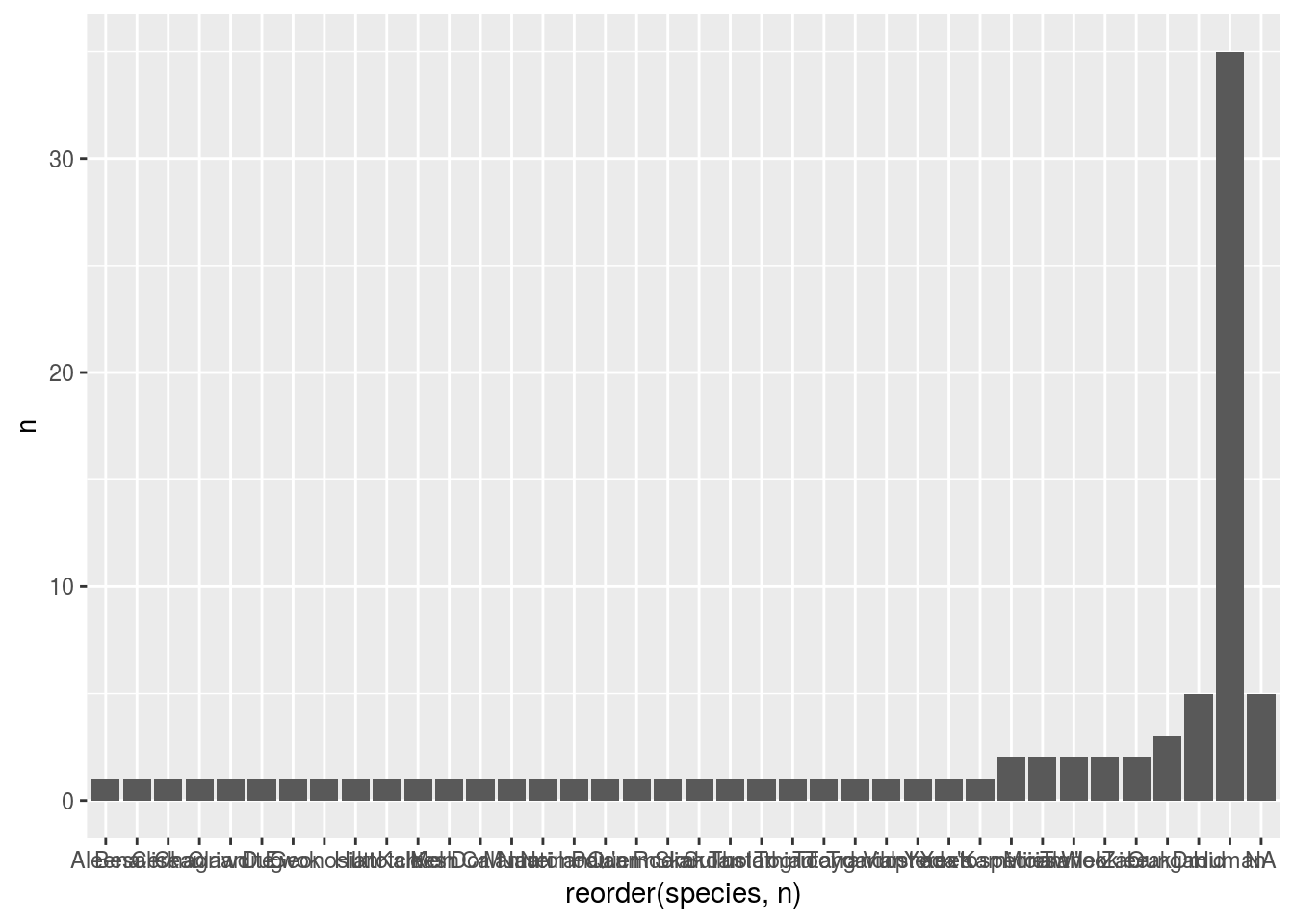
sw2 <- sw %>% count(species) %>%
mutate(species = fct_reorder(species, n))
ggplot(sw2, aes(x = reorder(species, n), y = n)) + geom_bar(stat = "identity")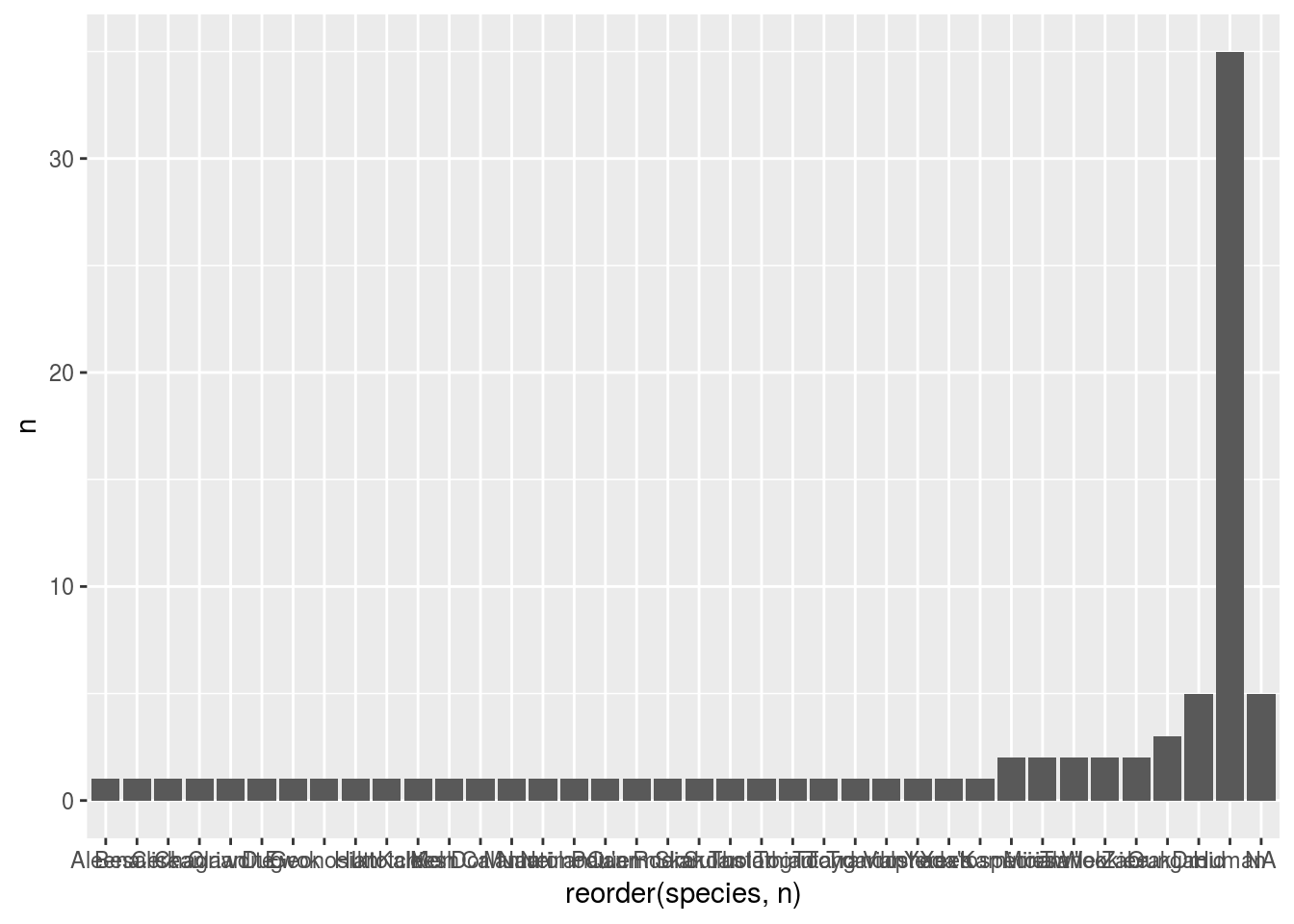
2 Luento 4: Erilaiset visualisoinnit ja analyysiprosessin kokonaisuus
2.1 Datojen vieminen toisiin ohjelmiin
Lyhyenä kertauksena vielä miten voit tuoda datoja R:ään ja viedä datojen R:stä toisiin ohjelmiin. Tähän mennessä olemme tuoneet datoja haven-paketin funktioilla, readr::read_csv()-funktiolla sekä readxl::read_excel()-funktiolla. Nämä paketit tarjoavat kirjoittamiseen vastaavat funktiot, jotka havenissa ovat haven::write_dta() jne. readr-paketilla kirjoitat .csv-datan readr::write_csv()-funktiolla. Excel-tiedoston voit kirjoittaa funktiolla writexl::write_xlsx().
Yleensä varmin tapa siirtää data R:stä SAS:iin, Stata:an tai SPSS:ään on a) tallentaa data ensin .csv-muotoon ja lukea se sitten ko. ohjelmaan ns. tekstinä. Kussakin ohjelmassa on oma tapansa tekstidatojen tuomiselle ja manuaalit/google on tässä hyvä apu.
Mikäli näiden eri pakettien muistaminen tuntuu työläältä kannattaa tututustua myös pakettiin rio: A Swiss-Army Knife for Data I/O. Paketissa on käytänössä kaksi funktiota import() ja export() ja funktiot arvaa luettavan/kirjoitettavan tiedoston päätteestä mitä pakettia käyttää. Eli jos haluat lukea vaikkapa Statan formaattia tai .csv-tiedoston, voit kirjoittaa vain näin:
ess <- rio::import(file = "./datasetit/ESS8e01.stata/ESS8e01.dta")
oecd <- rio::import(file = "./datasetit/qog_oecd_ts_jan18.csv")Jos taas haluat tallentaa dplyr::starwars-datan exceliksi ja csv:ksi, voit tehdä sen
sw <- dplyr::starwars
rio::export(x = sw, file = "./datasetit/starwars.xlsx") # excel
rio::export(x = sw, file = "./datasetit/starwars.csv") # csv2.2 Visualisoinnit
Seuraavassa käydään läpi aluksi ggplot2-kuvien ulkoasun räätälöimistä ja lopuksi kuvien tallentamista levylle.
2.2.1 Kuvien ulkoasun räätäloiminen
Tässä on yksinkertainen demo yhden kuvan räätälöimisestä. Ennen sen läpikäymistä tutustu kurssikurjoita lukuun 8 Refine your plots!
Likipitäen kaikia ggplot2-kuvien elementtejä on mahdollista muokata. Helpoin tapa päästä alkuun on ottaa joku valmis teema ja viilata sitä omien/kollegoiden/lehden vaatimuksien mukaiseksi.
Teen seuraavassa tuttuun starwars-dataan pari uutta muuttujaa ja ehostan kuvaa askel askeleelta.
library(tidyverse)
# luodaan data ja poistetaan Jabba
sw <- dplyr::starwars %>% filter(name != "Jabba Desilijic Tiure") %>%
# tehdään syntymävuosiluokkamuuttuja
mutate(birth_year_class = case_when(
birth_year <= 34 ~ "the old",
birth_year > 34 & birth_year <= 70 ~ "the middle aged",
birth_year > 70 ~ "the young",
TRUE ~ "not born"
),
# Tehdään muuttujasta faktori!
birth_year_class = factor(birth_year_class, levels = c("the young",
"the middle aged",
"the old",
"not born")),
# tehdään mys painoindeksi
bmi = mass / (height/100)^2) %>%
arrange(bmi) %>%
slice(1:25)Askel 1: Peruskuva
Tehdään hajontakuvio jossa kaikki uudet muuttujat näytillä!
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name))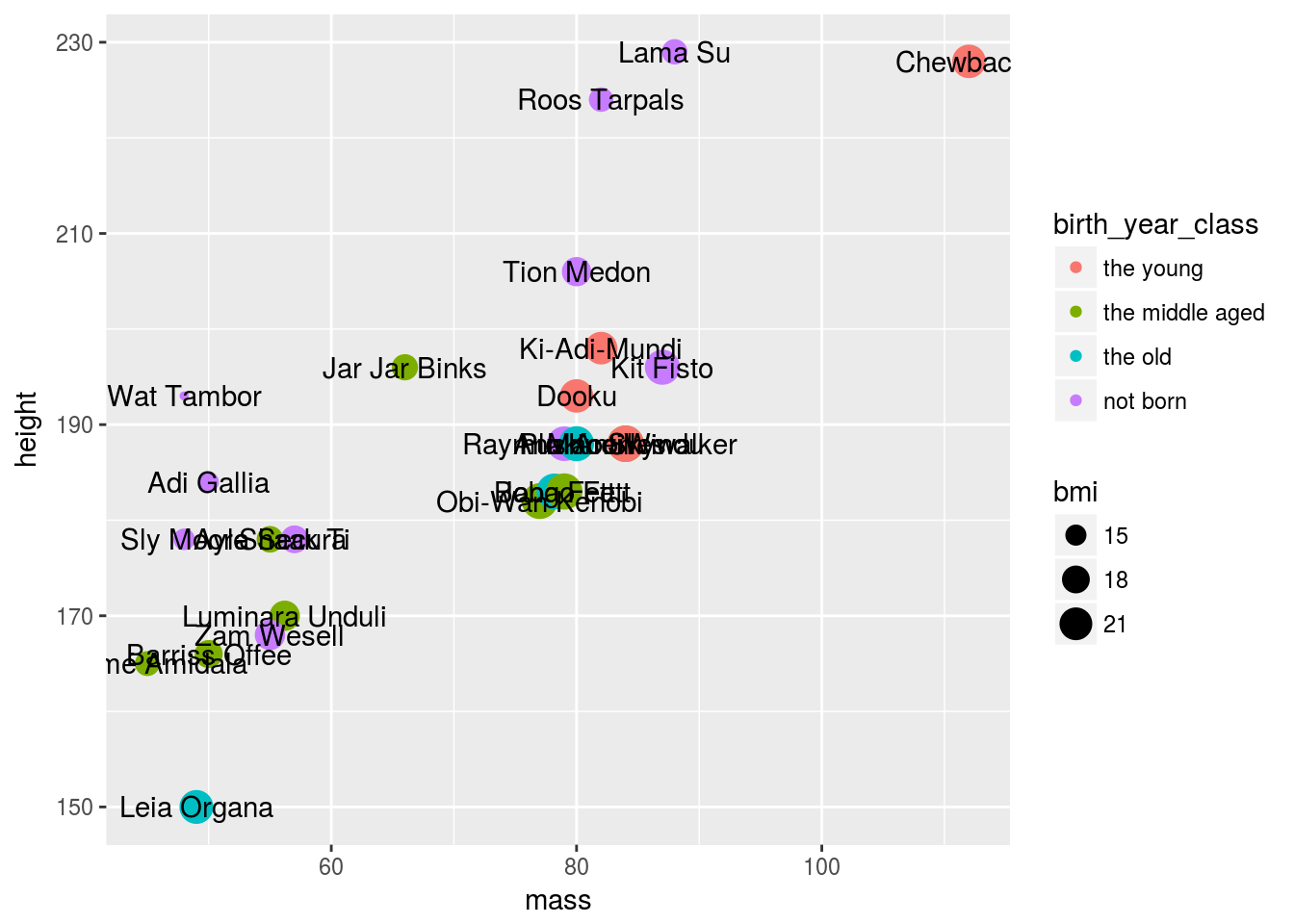
Askel 2: Valmiit teemat
ggplot2-paketissa on mukana 10 teemaa, jotka saat käyttöön lisäämällä theme_teemanimi()-kerroksen kuvaan. Voit antaa suoraan kullekin teemalle argumenteiksi base_size eli teeman fonttikoon sekä base_family eli teeman fontin. Tässä vaiheessa lisään vain pelkän teeman. Otsikot ovat jo tuttu asia ja lisään tässä vaiheessa kuvaan myös otsikkokerroksen funktiolla labs()
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name)) +
# Teema
theme_minimal() +
# Kuvan otsikot
labs(title = "Starwars otusten kehonkoostumus ja elinvuodet",
subtitle = "Esimerkkikuva ggplot2:n perusominaisuuksista",
size = "Painoindeksi",
color = "Luokiteltu ikä",
x = "Paino (kg)",
y = "Pituus (cm)",
caption = "data: dplyr::starwars")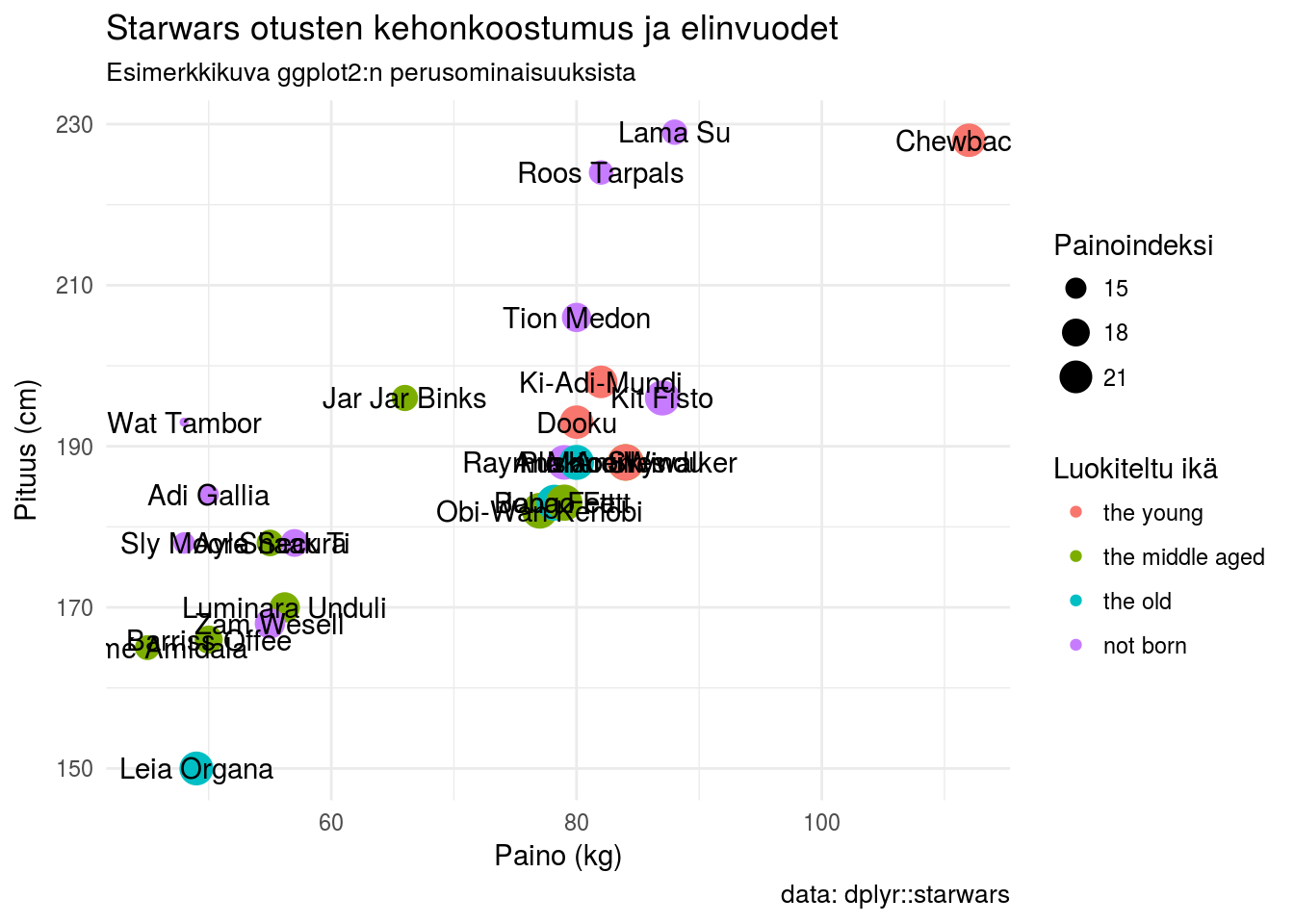
Askel 3: Värit
Riittävän hyvän väripaletin löytäminen kulloiseenkin tarkoitukseen on usein aikaavievä ja hermoja raastava vaihe. R:ssä on saatavilla muutama sata väriä jotka listattu mm. dokumenttiin Colors in R ja joita voi kutsua suoraan värin nimellä. ggplot2:ssa voit luoda omia väripaletteja funktioilla scale_color_manual() ja scale_fill_manual(). Funktiolla scale_fill_brewer(palette = "paletinnimi")/scale_color_brewer(palette = "paletinnimi") voit lisätä valmiita ColorBrewer-paletteja Alla kuvaan on lisätty manuaalinen väri palloille.
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name)) +
# Teema
theme_minimal() +
# Kuvan otsikot
labs(title = "Starwars otusten kehonkoostumus ja elinvuodet",
subtitle = "Esimerkkikuva ggplot2:n perusominaisuuksista",
size = "Painoindeksi",
color = "Luokiteltu ikä",
x = "Paino (kg)",
y = "Pituus (cm)",
caption = "data: dplyr::starwars") +
# lisätään oma väripaletti
scale_color_manual(values = c("royalblue", "springgreen", "red", "darkmagenta"))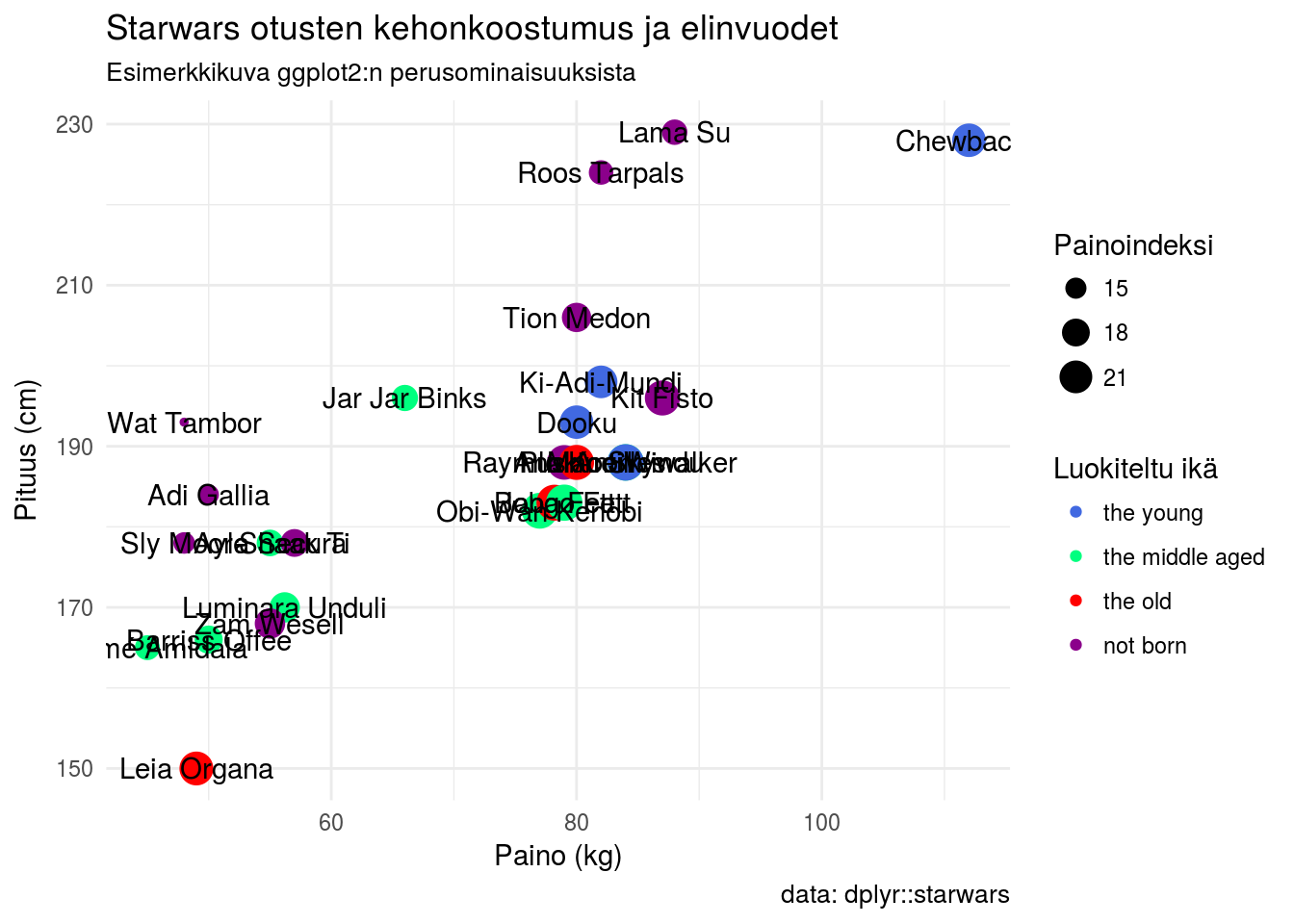
R:ään on olemassa useita valmiita väripaletteja sisältäviä paketteja. Emil Hvitfeldtin Comprehensive list of color palettes in r lienee kattavin yhteenveto paleteista. Itse käytän usein paketteja ggsci ja viridis. Kokeile vaikka ggsci::scale_color_futurama()
Askel 4: Fontit
Voit käyttää koneellesi asennettuja fontteja R:ssä extrafont-paketin avulla. Kun käytät paketettia ensimmäisen kerran, aja seuraavat komennot:
install.packages("extrafont") # asentaa paketin
library(extrafont) # lataa paketin
font_import() # tekee koneella olevista fonteista tietokannan paketin asennushakemistoonKun otat fontit myöhemmin käyttöön riittää kun ennen kuvan piirtoa aja komenot
library(extrafont)
loadfonts() # lataa fontit asennushakemiston tietokannastaSaat selville saatavilla olevat fontit komennolla extrafont::fonts(). Alla olevassa kuvassa olen antanut theme_minimal() funktiolle argumentiksi erään koneelleni asennetun fontin nimen Ubuntu. Fontin voi asentaa mm. google web fonts -palvelusta, mikäli sinulla on pääkäyttäjän oikeudet koneelle.
library(extrafont)
loadfonts()
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name)) +
# Teema
theme_minimal(base_family = "Ubuntu") +
# Kuvan otsikot
labs(title = "Starwars otusten kehonkoostumus ja elinvuodet",
subtitle = "Esimerkkikuva ggplot2:n perusominaisuuksista",
size = "Painoindeksi",
color = "Luokiteltu ikä",
x = "Paino (kg)",
y = "Pituus (cm)",
caption = "data: dplyr::starwars") +
# lisätään oma väripaletti
scale_color_manual(values = c("royalblue", "springgreen", "red", "darkmagenta"))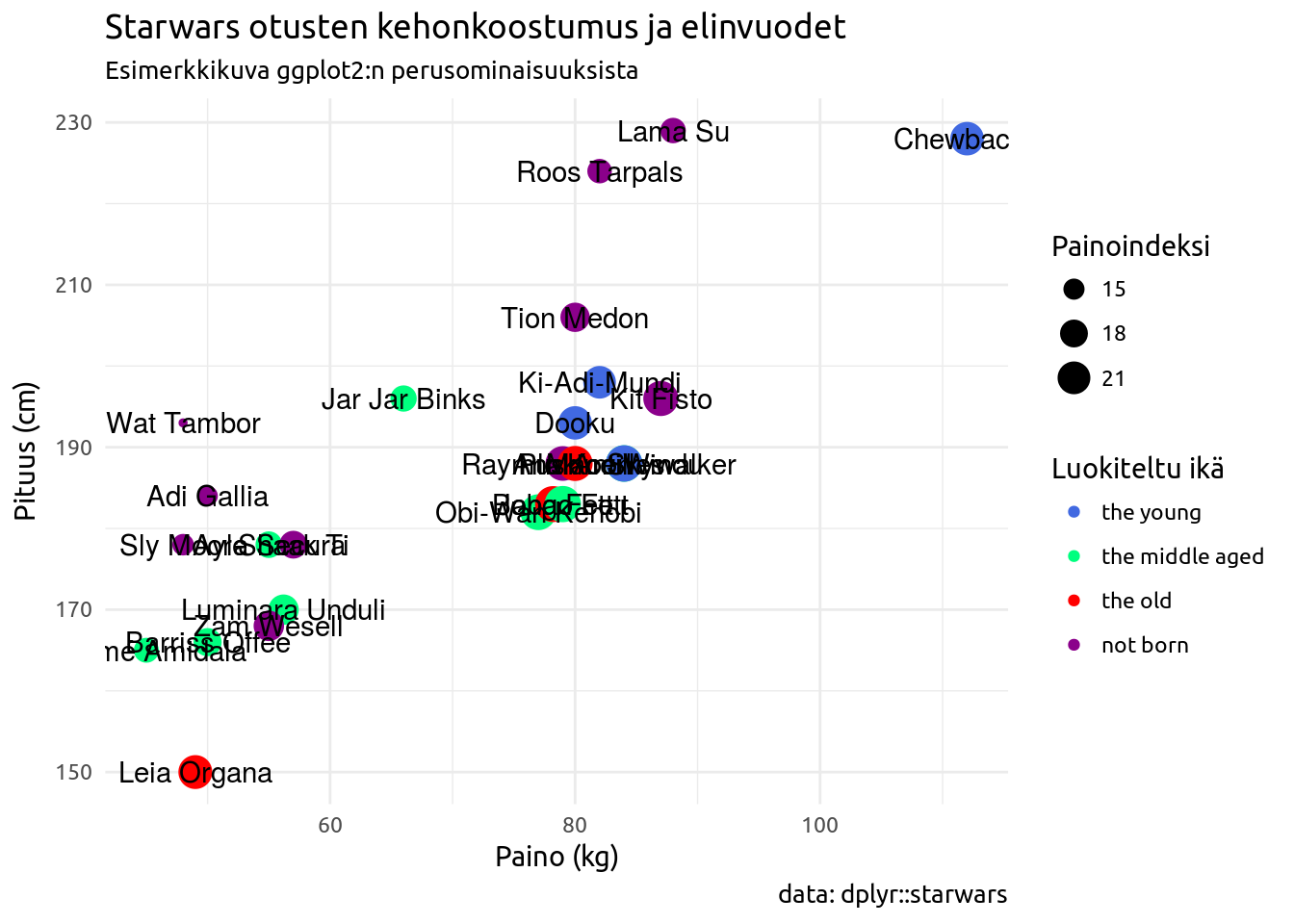
Askel 5: Teemojen kustomointi
Kuvan eri komponenteja muokataan funktion theme() avulla. Virallisen dokumentaation sivu Modify components of a theme kertoo kunkin muokattavan komponentin tyypin ja muokkausmahdollisuudet. Seuraavassa siiräämme selitteen alhaalle ja tasaamme akselin otsikot lähelle origoa.
library(extrafont)
loadfonts()
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name)) +
# Teema
theme_minimal(base_family = "Ubuntu") +
# Kuvan otsikot
labs(title = "Starwars otusten kehonkoostumus ja elinvuodet",
subtitle = "Esimerkkikuva ggplot2:n perusominaisuuksista",
size = "Painoindeksi",
color = "Luokiteltu ikä",
x = "Paino (kg)",
y = "Pituus (cm)",
caption = "data: dplyr::starwars") +
# lisätään oma väripaletti
scale_color_manual(values = c("royalblue", "springgreen", "red", "darkmagenta")) +
# Teeman kustomointi
theme(legend.position = "bottom",
axis.title = element_text(hjust = 0))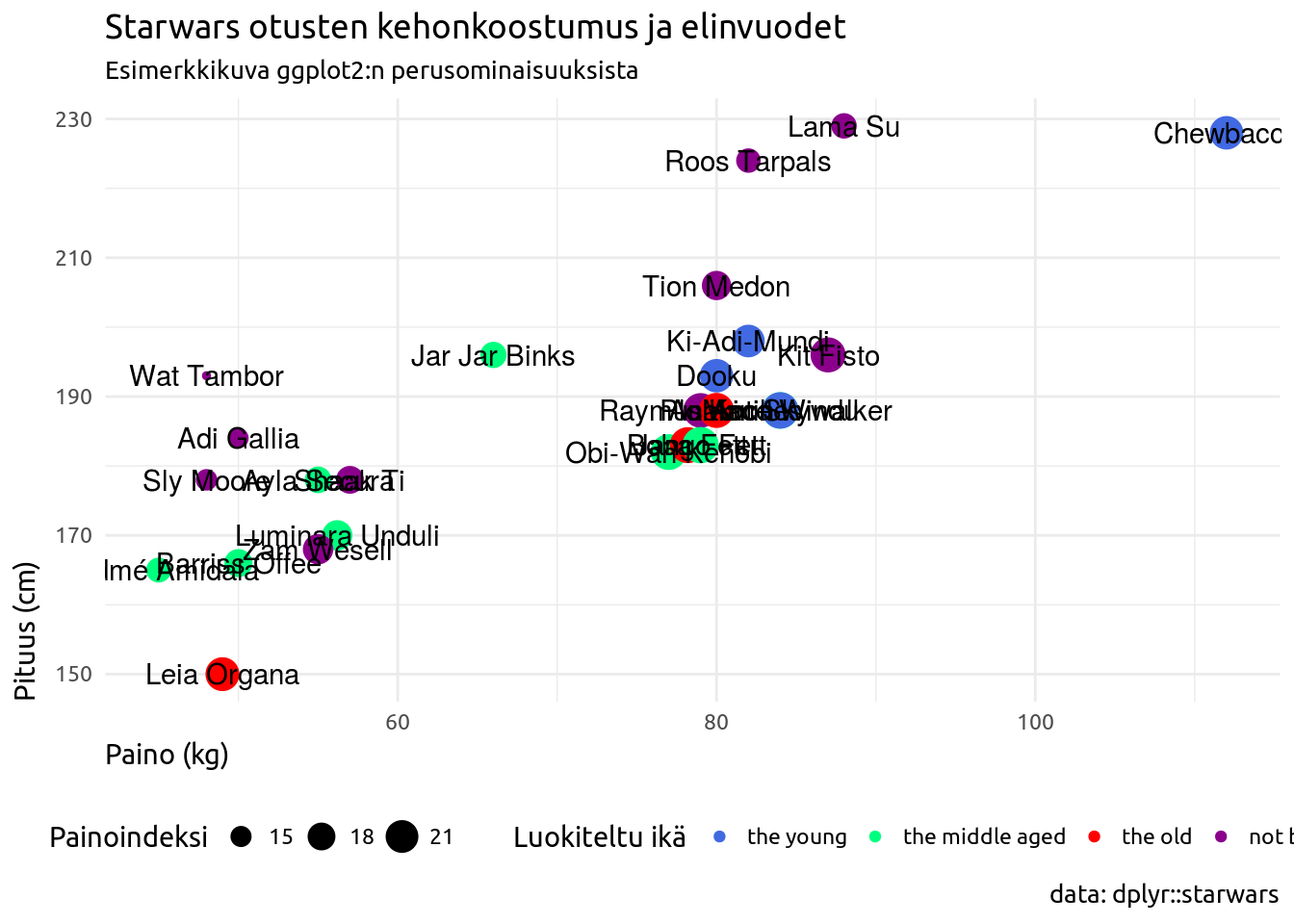
Askel 6: Tekstilabeleiden kustomointi
Tässä hajontakuviossa olen lisännyt pisteille myös kunkin hahmon nimen. Teeman fontin muokkaaminen ei muuta labeleiden fontteja, vaan se on tehtävä käsin. Seuraavassa muokkaa labeleiden fonttia ja nostan kutakin labelia hieman pisteen yläpuolelle. Huomaa että teen muutokset ~koodin puoliväliin
library(extrafont)
loadfonts()
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name), family = "Ubuntu", fontface = "bold", size = 3, nudge_y = 4) +
# Teema
theme_minimal(base_family = "Ubuntu") +
# Kuvan otsikot
labs(title = "Starwars otusten kehonkoostumus ja elinvuodet",
subtitle = "Esimerkkikuva ggplot2:n perusominaisuuksista",
size = "Painoindeksi",
color = "Luokiteltu ikä",
x = "Paino (kg)",
y = "Pituus (cm)",
caption = "data: dplyr::starwars") +
# lisätään oma väripaletti
scale_color_manual(values = c("royalblue", "springgreen", "red", "darkmagenta")) +
# Teeman kustomointi
theme(legend.position = "bottom",
axis.title = element_text(hjust = 0))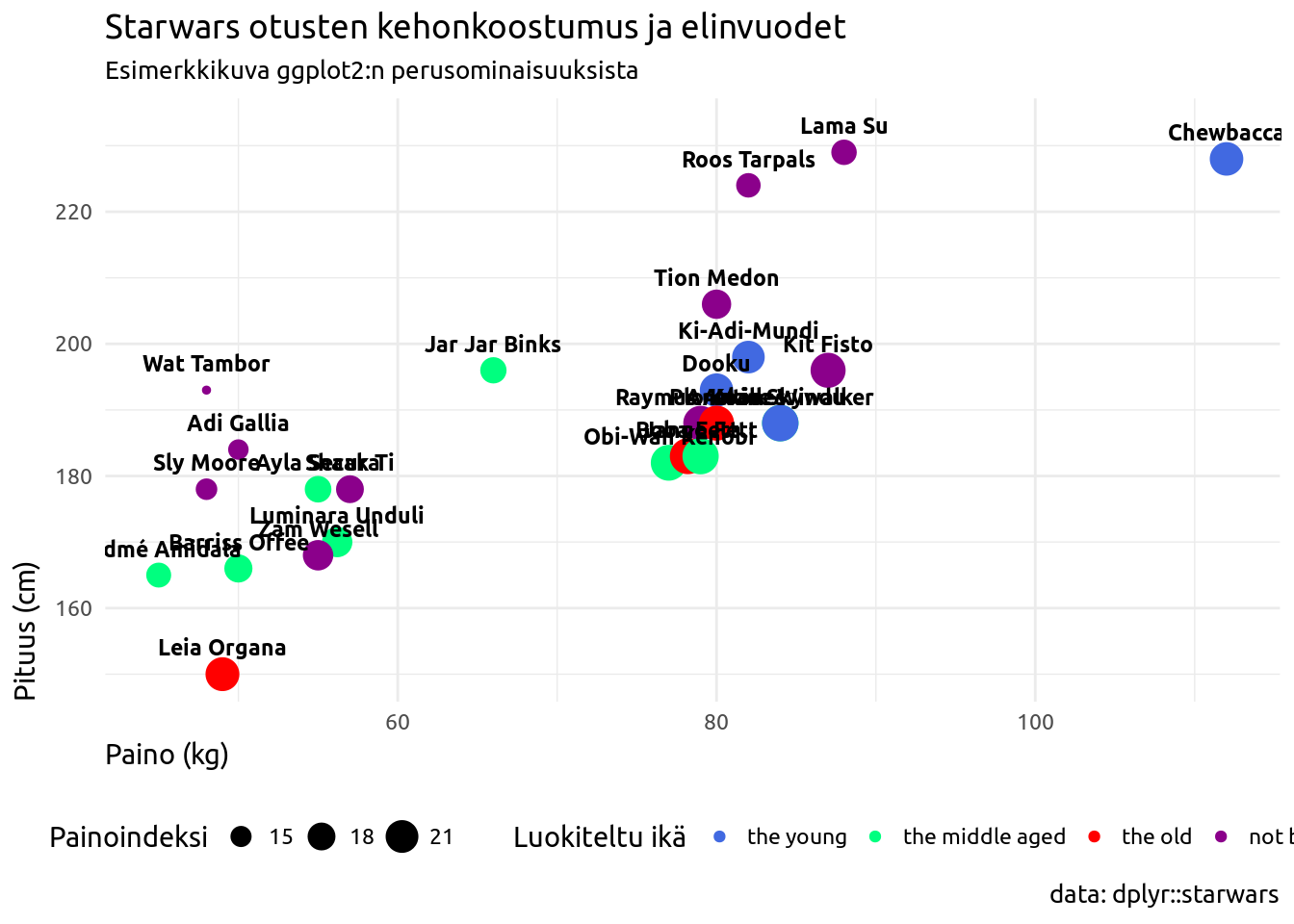
Askel 7: Päällekkäisen piirtämisen (overplotting) hallinta
Etenkin hajontakuvioissa, pisteet ja etenkin labelit menevät usein päällekkäin. Pisteiden päällekkäisyyttä voi ggplot2:ssa hoitaa “hajottamalla” pisteiden sijaintia käyttämällä geom_point():n sijaan geom_jitter():ia. Tässä tapauksessa riittää pelkkä läpinäkyvyyden lisääminen alpha argumentilla. Labeleiden päällekkäisyys hoidetaan asentamalla uusi paketti ggrepel. Alla kuva jossa sekä geom_point() että geom_text() kerrokset muokattu.
library(extrafont)
loadfonts()
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi), alpha = .6) +
# tekstille määritetään ainoastaan nimi
ggrepel::geom_text_repel(aes(label = name), family = "Ubuntu", fontface = "bold", size = 3, nudge_y = 4) +
# Teema
theme_minimal(base_family = "Ubuntu") +
# Kuvan otsikot
labs(title = "Starwars otusten kehonkoostumus ja elinvuodet",
subtitle = "Esimerkkikuva ggplot2:n perusominaisuuksista",
size = "Painoindeksi",
color = "Luokiteltu ikä",
x = "Paino (kg)",
y = "Pituus (cm)",
caption = "data: dplyr::starwars") +
# lisätään oma väripaletti
scale_color_manual(values = c("royalblue", "springgreen", "red", "darkmagenta")) +
# Teeman kustomointi
theme(legend.position = "bottom",
axis.title = element_text(hjust = 0))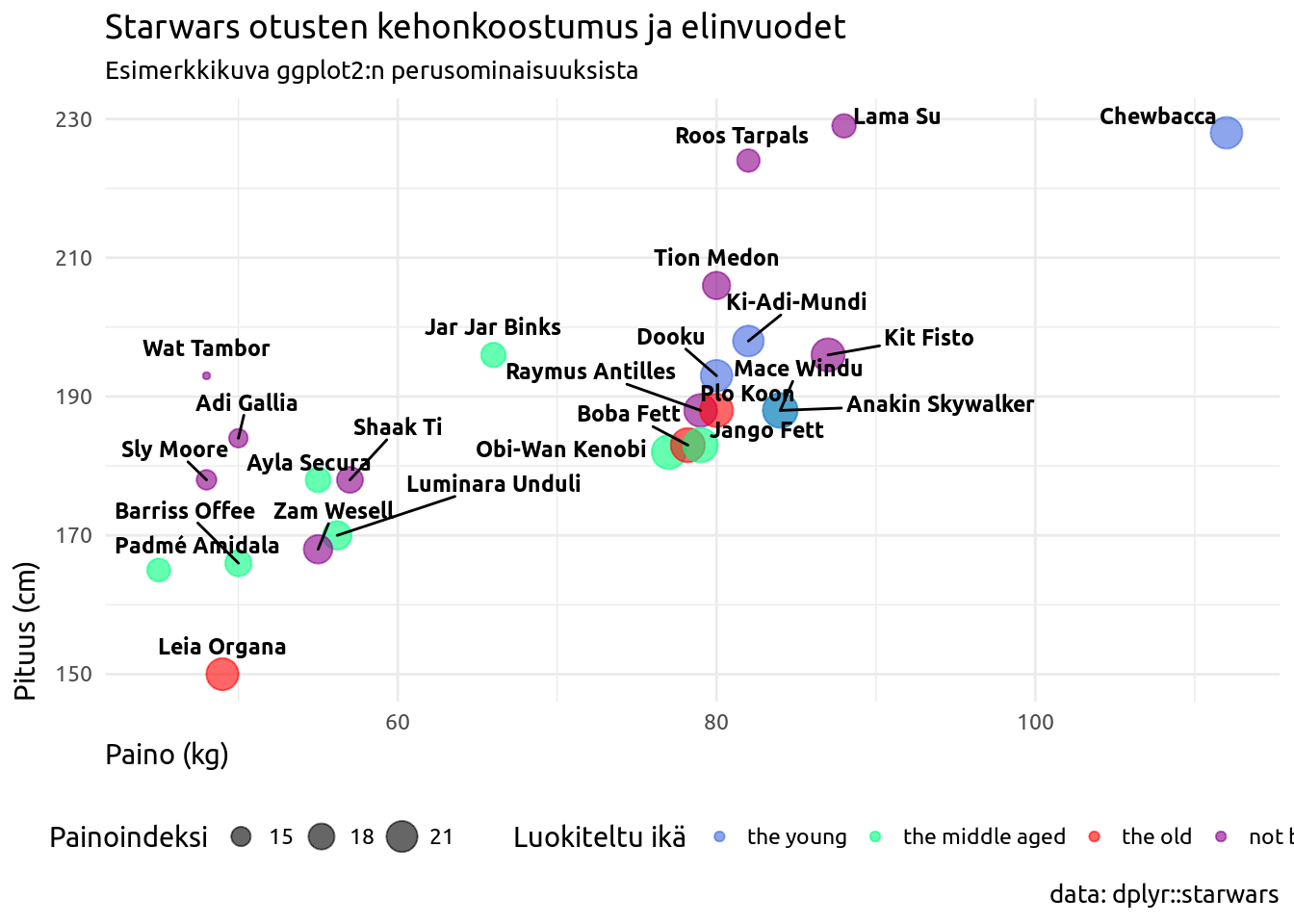
2.2.2 Lyhyt johdanto grafiikkaformaatteihin
Tilastografiikat tekemisessä on tärkeää tietää perusasiat eri grafiikkaformaateista. Grafiikkaformaattien kaksi päälinjaa ovat bittikarttagrafiikka ja vektorigrafiikka, joiden ominaisuudet tiivistetty ao. taulukkoon. Lue myös yo. lyhyet wikipedia-artikkelit mikäli ero ei ole aivan selvä.
Vektorigrafiikka vs. bittimappigrafiikka
| bittikartta (bitmap) | vektori | |
|---|---|---|
| tiedostopääte | .jpg, .png, .gif | .eps, .pdf, .svg, .ai |
| esimerkiksi | digikuva | googlen kartat |
| koostuu | miljoonista pikseleistä | pisteistä, viivoista ja polygoneista |
| tiedostokoko | suuri | pieni |
| muokkausohjelmisto | Gimp (Photoshop) | Inkscape (Illustrator) |
| sopii | verkoon, printtiin (korkearesoluutioisena) | printtiin, jälkikäsittelyyn, -svg-muodossa myös verkkoon |
Vektorigrafiikasta voi tehdä bittimappigrafiikkaa, mutta ei toisin päin #’ R:ssä tehtyjä kuvia voit tallentaa useisiin erilaisiin sekä vektori- että bittikarttaformaattiehin.
Staattinen vs. vuorovaikutteinen grafiikka
Bittikartta- ja vektorigrafiikka ovat ensisijaisesti ns. staattista grafiikka, jossa ei ole vuorovaikutteisia ominaisuuksia. “Tutkimusviestinnässä” perinteisen printattavan tutkimusraportin ohella yleistyy erilaiset verkkototeutukset kuten blogit tai verkkosovellukset. Kun sisältö on palvelimella ja lukeminen tapahtuu selaimella, niin kaikenlaiset verkkoteknologiat ovat käytettävissä. Verkkototeutusten kaksi päälinjaa, vuorovaikutteinen grafiikka & verkkosovellukset, on vedetty yhteen alla olevaan taulukkoon.
| grafiikan tyyppi | pros | cons |
|---|---|---|
| interaktiiviset kuviot | teknologian kehitys nopeaa | lyhyt elinkaari päivittyvien riippuvuuksien myötä |
| paljon vaihtoehtoja | hyvin sekava skene | |
| tarvitsee verkkoyhteydet | ||
| ei voi printata! | ||
| verkkosovellukset | hyvin joustavia | hostaus ja ylläpito |
| pystyy kaikkeen mihin R & linux | vaatii paljon räätälöintia |
Vuorovaikutteiset kuviot
Helpoin tapa tehdä ggplot2-kuvista vuorovaikutteisia on asentaa plotly()-paketti.
kuvaobjekti <- ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name))
plotly::ggplotly(kuvaobjekti)Lisää tietoa erilaisista vuorovaikutteisen grafiikan paketeista löytyy sivulta htmlwidgets - Showcase.
verkkosovellukset
shiny-paketin avulla voit tehdä vuorovaikutteisia analyysisovelluksia nettiselaimeen. Sovellukset ovat käteviä uuteen dataan tutustuttaessa, mutta shinylla voi tehdä myös monimutkaisempia sovelluksia ja niitä voi jakaa joko shinyapps.io:n tai omalle palvelimelle asennettavan shiny server:in kautta.
Tein pienen demon, jonka voit ladata itsellesi seuraavasti
# 1. päivitä kurssin paketti
devtools::install_git("https://gitlab.com/muuankarski/utur2018") # kurssin oma paketti
# 2. hae shiny komennolla
utur2018::lataa_shiny()
# 3. avaa sovellus muokattavaksi
file.edit("./app.R")
# 4. paina muokkausikkunan oikeasta yläkulmasta "Run App" -nappulaa!2.2.3 Kuvien tallentaminen levylle
Rstudiossa voit valita Plots-paneelista Export ja tallentaa kuvan Save as image dialogista kuuteen eri formaattiin:
- PNG - bittikartta-formaatti - tukee läpinäkyvyyttä - varma valinta verkkoon
- JPEG/JPG) - bittikartta-formaatti - pakattavissa pieneen tilaan - varma valinta verkkoon
- TIFF - bittikartta-formaatti - pakkaamaton, korkearesoluutioisena printtiin
- BMP - bittikartta-formaatti - perinteinen pakkaamaton formaatti. katoamassa.
- SVG - scalable vector graphics - avoin vektorigrafiikkamuoto. Uudet selaimet tukevat. Inkscape-grafiikkaohjelman oletusmuoto
- EPS - Encapsulated PostScript -
sekä pdf-formaattiin kohdasta “Save as PDF”.
pdf-formaatti on nykyisellään parhaiten yhteensopiva vektorigrafiikan muoto. Suurin haaste on eri fonttien käyttö
ggplot2-kuvien tallentaminen eri formaatteihin
ggplot2-paketilla tehtyjä kuvia voi tallentaa Rstudio-käyttöliittymästä käsin samoin kuin kaikkia muitakin formaatteja. Tallentaminen on kuitenkin näppärämpää kirjoittaa kuvion koodin yhteyteen ´ggsave()-funktiota käyttäen. ´ggsave()-funktio ymmärtää kuvion nimen päätteestä, mihin formaattiin kuva tallennetaan. Jos teet kuvion alla olevalla koodilla
kuvaobjekti <- ggplot(data=cars, aes(x=speed,y=dist)) + geom_point()
Voit tallentaa sen eri formaatteihin koodilla
ggsave(plot=kuvaobjekti, filename = "kuva.png")- bittimappi pngggsave(plot=kuvaobjekti, filename = "kuva.pdf")- vektori pdfggsave(plot=kuvaobjekti, filename = "kuva.svg")- vektori svg
Lisäparametreinä ´ggsave()`-funktioon voi laittaa mm. seuraavat
filenameFile name to create on disk.plotPlot to save, defaults to last plot displayed.deviceDevice to use (function or any of the recognized extensions, e.g. “pdf”). By default, extracted from filename extension. ggsave currently recognises eps/ps, tex (pictex), pdf, jpeg, tiff, png, bmp, svg and wmf (windows only).pathPath to save plot to (combined with filename).scaleMultiplicative scaling factor.width,heightPlot dimensions, defaults to size of current graphics device.unitsUnits for width and height when specified explicitly (in, cm, or mm)dpiResolution used for raster outputs.limitsizeWhen TRUE (the default), ggsave will not save images larger than 50x50 inches, to prevent the common error of specifying dimensions in pixels.
Mikäli et ole vielä luonut kansiota kotitehtava4/kuviot luo se komennolla dir.create(path="./kotitehtava4/kuviot", recursive=TRUE, showWarnings = FALSE)
Piirrä kuvat koodilla kuva <- ggplot(data=cars, aes(x=speed,y=dist)) + geom_point()` ja kirjoita miten se tallennetaan ko. kansioon png, svg, tiff, jpg, eps, pdf ja bmp muodoissa.
Avaan kaikki kuvat koneellasi ja tarkastele niitä!
2.2.4 Kartat
Perinteisen tilastollisen ohjelmsiston lisäksi R:ssä on erinomaiset spatiaalisen datan käsittelyn ja analysoinnin valmiudet. Internet on pullollaan erilaisia tutoriaaleja karttojen tekemisestä R:llä, mutta kokonaisuus on liian laaja tällä kurssilla käsiteltäväksi linkitän tähän vain pari hyvää resurssia joilla pääsee alkuun:
- Kurssikirjasta luku 7 Draw maps
- Euroopan maiden karttoja Eurostatin datoilla
- Pari omaa viimeistellympää demoa Eurostatin datoista Eurostat (regional) maps using R and eurostat-package
2.3 Analyysiprosessin kokonaisuus
Data-analyysiprosessin kokonaisuuden hallintaan ei ole olemassa yhtä oikeaa vaihtoehtoa. Oikea projektin jäsentämisen ja tiedonhallinnan malli riippuu aina tehtävästä ja tekijöistä. Kaksi välinettä 1) Rstudion projektit ja 2) Rmarkdown ovat kuitenkin ehdottoman hyödyllisiä. Projekteista on kurssin aikana jo puhuttu, samoin Rmarkdownista, mutta tutustu jälkimmäiseen paremmin vielä alla olevan luennon pohjalta.
Luennon ohella perehdy myös tähän artikkeliin Wilson G, Bryan J, Cranston K, Kitzes J, Nederbragt L, Teal TK (2017) Good enough practices in scientific computing. PLoS Comput Biol 13(6): e1005510.. Siinä käydään läpi hyvän data-analyysin peruskäsitteitä ja ehdotetaan myös yhtä melko hyvää tiedonhallinnan mallia.
2.3.1 Rmarkdown tilastollisten raporttien kirjoittamisessa
Getting started with R Markdown from RStudio, Inc. on Vimeo.
Rmarkdownin laajennokset
2017-2018 Markus Kainu.

Tämä teos on lisensoitu Creative Commons Nimeä 4.0 Kansainvälinen -lisenssillä.