Erilaiset visualisoinnit ja analyysiprosessi kokonaisuus
Kertausta
Datan käsittely
Taulukot
Yksinkertaiset taulukot
library(tidyverse)
sw <- dplyr::starwars %>%
# poistetaan NA:t muuttujista species, gender
filter(!is.na(species),
!is.na(gender))
# yhden muuttujan luokkien frekvenssit
tbl <- as.data.frame(
table(sw$species, useNA = "ifany")
)
head(tbl, 10)## Var1 Freq
## 1 Aleena 1
## 2 Besalisk 1
## 3 Cerean 1
## 4 Chagrian 1
## 5 Clawdite 1
## 6 Droid 2
## 7 Dug 1
## 8 Ewok 1
## 9 Geonosian 1
## 10 Gungan 3# OR
sw %>% count(species)## # A tibble: 37 x 2
## species n
## <chr> <int>
## 1 Aleena 1
## 2 Besalisk 1
## 3 Cerean 1
## 4 Chagrian 1
## 5 Clawdite 1
## 6 Droid 2
## 7 Dug 1
## 8 Ewok 1
## 9 Geonosian 1
## 10 Gungan 3
## # … with 27 more rows# yhden muuttujan luokkien osuudet
tbl <- as.data.frame(
prop.table(table(sw$species, useNA = "ifany"))*100
)
head(tbl, 10)## Var1 Freq
## 1 Aleena 1.265823
## 2 Besalisk 1.265823
## 3 Cerean 1.265823
## 4 Chagrian 1.265823
## 5 Clawdite 1.265823
## 6 Droid 2.531646
## 7 Dug 1.265823
## 8 Ewok 1.265823
## 9 Geonosian 1.265823
## 10 Gungan 3.797468# OR
sw %>% count(species) %>%
mutate(share = n/sum(n)*100) %>%
arrange(desc(share))## # A tibble: 37 x 3
## species n share
## <chr> <int> <dbl>
## 1 Human 35 44.3
## 2 Gungan 3 3.80
## 3 Droid 2 2.53
## 4 Kaminoan 2 2.53
## 5 Mirialan 2 2.53
## 6 Twi'lek 2 2.53
## 7 Wookiee 2 2.53
## 8 Zabrak 2 2.53
## 9 Aleena 1 1.27
## 10 Besalisk 1 1.27
## # … with 27 more rows# kahden muuttujan luokkien frekvenssit
tbl <- as.data.frame(
table(sw$species,sw$gender, useNA = "ifany")
)
head(tbl, 10)## Var1 Var2 Freq
## 1 Aleena female 0
## 2 Besalisk female 0
## 3 Cerean female 0
## 4 Chagrian female 0
## 5 Clawdite female 1
## 6 Droid female 0
## 7 Dug female 0
## 8 Ewok female 0
## 9 Geonosian female 0
## 10 Gungan female 0# Or
sw %>%
group_by(species,gender) %>%
count() %>%
ungroup() %>%
group_by(gender) %>%
mutate(osuus = n/sum(n)*100) %>%
ungroup() %>%
arrange(gender,desc(osuus))## # A tibble: 40 x 4
## species gender n osuus
## <chr> <chr> <int> <dbl>
## 1 Human female 9 56.2
## 2 Mirialan female 2 12.5
## 3 Clawdite female 1 6.25
## 4 Kaminoan female 1 6.25
## 5 Tholothian female 1 6.25
## 6 Togruta female 1 6.25
## 7 Twi'lek female 1 6.25
## 8 Hutt hermaphrodite 1 100
## 9 Human male 26 43.3
## 10 Gungan male 3 5
## # … with 30 more rowsMuuttujien uudelleenluokittelu
Jatkuvan pilkkominen luokkiin
sw$pituus_lk[sw$height < 100] <- "alle metrin"
sw$pituus_lk[sw$height >= 100 & sw$height < 150] <- "metristä puoleentoista"
sw$pituus_lk[sw$height >= 150 & sw$height < 200] <- "puolestatoista kahteen metriin"
sw$pituus_lk[sw$height >= 200] <- "yli kaksi metriä"
sw$pituus_lk[is.na(sw$pituus_lk)] <- "ei pituutta datassa"
table(sw$pituus_lk, useNA = "ifany")##
## alle metrin ei pituutta datassa
## 4 5
## metristä puoleentoista puolestatoista kahteen metriin
## 3 56
## yli kaksi metriä
## 11sw <- sw %>%
mutate(height_lk = case_when(
height < 100 ~ "alle metrin",
height >= 100 & height < 150 ~ "metristä puoleentoista",
height >= 150 & height < 200 ~ "puolestatoista kahteen metriin",
height >= 200 ~ "yli kaksi metriä",
TRUE ~ "ei pituutta datassa"
))
sw %>% count(height_lk)## # A tibble: 5 x 2
## height_lk n
## <chr> <int>
## 1 alle metrin 4
## 2 ei pituutta datassa 5
## 3 metristä puoleentoista 3
## 4 puolestatoista kahteen metriin 56
## 5 yli kaksi metriä 11Ehdollinen luokittelu ifelse
sw$pituus_lk <- ifelse(sw$height < 100, # ehto
"alle metrin", # arvo jos ehto täyttyy
"vähintään metrin") # arvo jos ehto ei täyty
table(sw$pituus_lk, useNA = "ifany")##
## alle metrin vähintään metrin <NA>
## 4 70 5sw <- sw %>%
mutate(height_lk = ifelse(height < 100,
"alle metrin",
ifelse(height < 200,
"yli metrin, mutta alle kaks",
"kolmatta metriä")))
sw %>% count(height_lk)## # A tibble: 4 x 2
## height_lk n
## <chr> <int>
## 1 <NA> 5
## 2 alle metrin 4
## 3 kolmatta metriä 11
## 4 yli metrin, mutta alle kaks 59Luokkien uudelleenluokittelu
# Alkuperäinen luokittelu
sw$pituus_lk[sw$height < 100] <- "alle metrin"
sw$pituus_lk[sw$height >= 100 & sw$height < 150] <- "metristä puoleentoista"
sw$pituus_lk[sw$height >= 150 & sw$height < 200] <- "puolestatoista kahteen metriin"
sw$pituus_lk[sw$height >= 200] <- "yli kaksi metriä"
sw$pituus_lk[is.na(sw$pituus_lk)] <- "ei pituutta datassa"
table(sw$pituus_lk, useNA = "ifany")##
## alle metrin ei pituutta datassa
## 4 5
## metristä puoleentoista puolestatoista kahteen metriin
## 3 56
## yli kaksi metriä
## 11## Josta karkeampi luokittelu
sw$pituus_lk2[sw$pituus_lk %in% c("alle metrin","metristä puoleentoista")] <- "alle puolentoistametrin"
sw$pituus_lk2[!sw$pituus_lk %in% c("alle metrin","metristä puoleentoista")] <- "yli puolentoistametrin"
table(sw$pituus_lk2, useNA = "ifany")##
## alle puolentoistametrin yli puolentoistametrin
## 7 72# Alkuperäinen luokittelu
sw <- sw %>%
mutate(height_lk = case_when(
height < 100 ~ "alle metrin",
height >= 100 & height < 150 ~ "metristä puoleentoista",
height >= 150 & height < 200 ~ "puolestatoista kahteen metriin",
height >= 200 ~ "yli kaksi metriä",
TRUE ~ "ei pituutta datassa"
))
sw %>% count(height_lk)## # A tibble: 5 x 2
## height_lk n
## <chr> <int>
## 1 alle metrin 4
## 2 ei pituutta datassa 5
## 3 metristä puoleentoista 3
## 4 puolestatoista kahteen metriin 56
## 5 yli kaksi metriä 11## Josta karkeampi luokittelu
sw <- sw %>%
mutate(height_lk2 = case_when(
height_lk %in% c("alle metrin","metristä puoleentoista") ~ "alle puolentoistametrin",
!height_lk %in% c("alle metrin","metristä puoleentoista")~ "yli puolentoistametrin"
))
sw %>% count(height_lk2)## # A tibble: 2 x 2
## height_lk2 n
## <chr> <int>
## 1 alle puolentoistametrin 7
## 2 yli puolentoistametrin 72Jatkuvan muuttujan luokittelu jakauman pohjalta
sw %>%
mutate(height_lk3 = cut_interval(x = height, n = 4)) %>%
select(name,height,height_lk3) %>%
count(height_lk3)## # A tibble: 5 x 2
## height_lk3 n
## <fct> <int>
## 1 [66,116] 5
## 2 (116,165] 11
## 3 (165,214] 52
## 4 (214,264] 6
## 5 <NA> 5Säännölliset lausekkeet eli regular expressions
- http://stat545.com/block022_regular-expression.html
- Introduction to stringr https://cran.r-project.org/web/packages/stringr/vignettes/stringr.html
library(tidyverse)
## ------------------------------------------------------------------------
sw <- dplyr::starwars
# str(sw)
# summary(sw)
## ------------------------------------------------------------------------
head(sw)## # A tibble: 6 x 13
## name height mass hair_color skin_color eye_color birth_year gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
## 1 Luke… 172 77 blond fair blue 19 male
## 2 C-3PO 167 75 <NA> gold yellow 112 <NA>
## 3 R2-D2 96 32 <NA> white, bl… red 33 <NA>
## 4 Dart… 202 136 none white yellow 41.9 male
## 5 Leia… 150 49 brown light brown 19 female
## 6 Owen… 178 120 brown, gr… light blue 52 male
## # … with 5 more variables: homeworld <chr>, species <chr>, films <list>,
## # vehicles <list>, starships <list>## Filttreoidaan arvoilla
sw %>% filter(species == "Human")## # A tibble: 35 x 13
## name height mass hair_color skin_color eye_color birth_year gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
## 1 Luke… 172 77 blond fair blue 19 male
## 2 Dart… 202 136 none white yellow 41.9 male
## 3 Leia… 150 49 brown light brown 19 female
## 4 Owen… 178 120 brown, gr… light blue 52 male
## 5 Beru… 165 75 brown light blue 47 female
## 6 Bigg… 183 84 black light brown 24 male
## 7 Obi-… 182 77 auburn, w… fair blue-gray 57 male
## 8 Anak… 188 84 blond fair blue 41.9 male
## 9 Wilh… 180 NA auburn, g… fair blue 64 male
## 10 Han … 180 80 brown fair brown 29 male
## # … with 25 more rows, and 5 more variables: homeworld <chr>,
## # species <chr>, films <list>, vehicles <list>, starships <list>## ------------------------------------------------------------------------
sw %>% filter(species %in% c("Human","Droid"))## # A tibble: 40 x 13
## name height mass hair_color skin_color eye_color birth_year gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
## 1 Luke… 172 77 blond fair blue 19 male
## 2 C-3PO 167 75 <NA> gold yellow 112 <NA>
## 3 R2-D2 96 32 <NA> white, bl… red 33 <NA>
## 4 Dart… 202 136 none white yellow 41.9 male
## 5 Leia… 150 49 brown light brown 19 female
## 6 Owen… 178 120 brown, gr… light blue 52 male
## 7 Beru… 165 75 brown light blue 47 female
## 8 R5-D4 97 32 <NA> white, red red NA <NA>
## 9 Bigg… 183 84 black light brown 24 male
## 10 Obi-… 182 77 auburn, w… fair blue-gray 57 male
## # … with 30 more rows, and 5 more variables: homeworld <chr>,
## # species <chr>, films <list>, vehicles <list>, starships <list>## ------------------------------------------------------------------------
rodut <- unique(sw$species)
### sisältää merkin 'n'
grep(pattern = "n", x = rodut) # indeksit## [1] 1 4 7 8 10 11 12 14 18 20 21 22 23 25 27 28 29 32 33 34 35 38grep(pattern = "n", x = rodut, value=TRUE) # arvot## [1] "Human" "Rodian" "Trandoshan" "Mon Calamari"
## [5] "Sullustan" "Neimodian" "Gungan" "Toydarian"
## [9] "Vulptereen" "Toong" "Cerean" "Nautolan"
## [13] "Tholothian" "Quermian" "Chagrian" "Geonosian"
## [17] "Mirialan" "Kaminoan" "Aleena" "Skakoan"
## [21] "Muun" "Pau'an"grepl(pattern = "n", x = rodut) # looginen vektori (alkuperäisen pituinen) siitä täyttyykö ehto## [1] TRUE FALSE FALSE TRUE FALSE FALSE TRUE TRUE FALSE TRUE TRUE
## [12] TRUE FALSE TRUE FALSE FALSE FALSE TRUE FALSE TRUE TRUE TRUE
## [23] TRUE FALSE TRUE FALSE TRUE TRUE TRUE FALSE FALSE TRUE TRUE
## [34] TRUE TRUE FALSE FALSE TRUE### päättyy merkkiin 'k'
grep("k$", rodut, value=TRUE)## [1] "Ewok" "Zabrak" "Twi'lek" "Besalisk"### alkaa joko merkillä N/n tai M/n
grep("^N|^m", rodut, value=TRUE, ignore.case = TRUE)## [1] "Mon Calamari" "Neimodian" "Nautolan" "Mirialan"
## [5] "Muun"# filtteroi rivit regular expressioilla
sw[grepl("k$", sw$species),c("name","species")]## # A tibble: 6 x 2
## name species
## <chr> <chr>
## 1 Wicket Systri Warrick Ewok
## 2 Darth Maul Zabrak
## 3 Bib Fortuna Twi'lek
## 4 Ayla Secura Twi'lek
## 5 Eeth Koth Zabrak
## 6 Dexter Jettster Besalisk# Or
sw %>% filter(grepl("k$", species)) %>% select(name,species)## # A tibble: 6 x 2
## name species
## <chr> <chr>
## 1 Wicket Systri Warrick Ewok
## 2 Darth Maul Zabrak
## 3 Bib Fortuna Twi'lek
## 4 Ayla Secura Twi'lek
## 5 Eeth Koth Zabrak
## 6 Dexter Jettster Besalisk# valitse muuttujat regular expressioilla
sw[,grep("color$", names(sw), value=TRUE)]## # A tibble: 87 x 3
## hair_color skin_color eye_color
## <chr> <chr> <chr>
## 1 blond fair blue
## 2 <NA> gold yellow
## 3 <NA> white, blue red
## 4 none white yellow
## 5 brown light brown
## 6 brown, grey light blue
## 7 brown light blue
## 8 <NA> white, red red
## 9 black light brown
## 10 auburn, white fair blue-gray
## # … with 77 more rowssw %>% select(
grep("color$", names(.), value=TRUE)
)## # A tibble: 87 x 3
## hair_color skin_color eye_color
## <chr> <chr> <chr>
## 1 blond fair blue
## 2 <NA> gold yellow
## 3 <NA> white, blue red
## 4 none white yellow
## 5 brown light brown
## 6 brown, grey light blue
## 7 brown light blue
## 8 <NA> white, red red
## 9 black light brown
## 10 auburn, white fair blue-gray
## # … with 77 more rowsSotkuisten arvojen siivoaminen
luo_kohinaa <- function(n, selection = letters, length = 5){
apply(replicate(n, sample(c(selection, ""), length, replace=TRUE, prob=c(rep(0.7/length(selection), length(selection)), .3))), 2, paste0, collapse="")
}
n <- 10
noisystring <- paste0(luo_kohinaa(n),
sample(1990:2015, n, rep=T),
luo_kohinaa(n))
noisystring## [1] "szv2010gsrt" "prin2004oco" "ohwm2001yzzzb" "okquc1995uki"
## [5] "ivsw1993cjzuy" "qbyak1995wt" "yavr1996zotz" "vsigf2011wp"
## [9] "brh2006agts" "wyzap2000gkye"## Korvataan kirjaimet tyhjällä (jää vain numerot)
gsub(pattern = "[a-zA-Z]", replacement = "", noisystring)## [1] "2010" "2004" "2001" "1995" "1993" "1995" "1996" "2011" "2006" "2000"n <- 10
selection <- c(letters, "&", "_")
noisystring2 <- paste0(luo_kohinaa(n, selection),
sample(1990:2015, n, rep=T),
luo_kohinaa(n, selection))
noisystring2## [1] "gpbb1992owyz" "rir2011cnom_" "sdho2011snjf" "iccw2001vj"
## [5] "mqtad2006uwi" "_ab2008vd" "gmzn2011zjeug" "ky2015pqv"
## [9] "xa_r2002k&pk" "phm1991mfgf"## Korvataan kaikki paitsi numerot
gsub("[^0-9]", "", noisystring2)## [1] "1992" "2011" "2011" "2001" "2006" "2008" "2011" "2015" "2002" "1991"# pilkotaan dataa
noisydata <- tibble::tibble(noisystring,noisystring2)
noisydata$noisydata_numbers <- gsub("[^0-9]", "", noisydata$noisystring2)
noisydata$noisydata_letters <- gsub("[0-9]", "", noisydata$noisystring2)
noisydata## # A tibble: 10 x 4
## noisystring noisystring2 noisydata_numbers noisydata_letters
## <chr> <chr> <chr> <chr>
## 1 szv2010gsrt gpbb1992owyz 1992 gpbbowyz
## 2 prin2004oco rir2011cnom_ 2011 rircnom_
## 3 ohwm2001yzzzb sdho2011snjf 2011 sdhosnjf
## 4 okquc1995uki iccw2001vj 2001 iccwvj
## 5 ivsw1993cjzuy mqtad2006uwi 2006 mqtaduwi
## 6 qbyak1995wt _ab2008vd 2008 _abvd
## 7 yavr1996zotz gmzn2011zjeug 2011 gmznzjeug
## 8 vsigf2011wp ky2015pqv 2015 kypqv
## 9 brh2006agts xa_r2002k&pk 2002 xa_rk&pk
## 10 wyzap2000gkye phm1991mfgf 1991 phmmfgf# Valitaan yli 2000 rivit
noisydata %>%
filter(noisydata_numbers > 2000) %>%
# ja vain väliviivan sisältävät sarakkeet
select(contains("_"))## # A tibble: 8 x 2
## noisydata_numbers noisydata_letters
## <chr> <chr>
## 1 2011 rircnom_
## 2 2011 sdhosnjf
## 3 2001 iccwvj
## 4 2006 mqtaduwi
## 5 2008 _abvd
## 6 2011 gmznzjeug
## 7 2015 kypqv
## 8 2002 xa_rk&pkFaktorit
Faktoreilla kuvataan kategorisia/järjestysasteikollisia muuttujia, jonka arvoilla on tietty järjestys. Faktoreita voi muodostaa joko funktioilla factor() tai readr::parse_factor().
x1 <- c("Joulu", "Huhti", "Tammi", "Maalis")
kk_levelit <- c(
"Tammi", "Helmi", "Maalis", "Huhti", "Touko", "Kesä",
"Heinä", "Elo", "Syys", "Loka", "Marras", "Joulu"
)
factor(x = x1, levels = kk_levelit)## [1] Joulu Huhti Tammi Maalis
## 12 Levels: Tammi Helmi Maalis Huhti Touko Kesä Heinä Elo Syys ... Joulureadr::parse_factor(x1, kk_levelit)## [1] Joulu Huhti Tammi Maalis
## 12 Levels: Tammi Helmi Maalis Huhti Touko Kesä Heinä Elo Syys ... JouluFaktorit ja starwars-data
library(tidyverse)
sw <- dplyr::starwars
# kirjainvektori
ggplot(sw, aes(x=name,y=height)) +
geom_point() +
theme(axis.text = element_text(angle=90))
# faktori
sw$name <- as.factor(sw$name) #
ggplot(sw, aes(x=name,y=height)) +
geom_point() +
theme(axis.text = element_text(angle=90))
# reorder
sw$name <- forcats::fct_reorder(.f = sw$name, .x = sw$height) #
ggplot(sw, aes(x=name,y=height)) +
geom_point() +
theme(axis.text = element_text(angle=90))
# relevel
# f <- factor(c("a", "b", "c"))
# fct_relevel(f)
# fct_relevel(f, "c")
# fct_relevel(f, "b", "a")
# reverse
sw$name <- forcats::fct_rev(f = sw$name) #
ggplot(sw, aes(x=name,y=height)) +
geom_point() +
theme(axis.text = element_text(angle=90))
sw$name <- forcats::fct_relevel(.f = sw$name, "Luke Skywalker") #
ggplot(sw, aes(x=name,y=height)) +
geom_point() +
theme(axis.text = element_text(angle=90))
Datojen yhdistäminen
Rivien pinoaminen dplyr::bind_rows()
library(tidyverse)
library(rqog)
oecd <- read_qog(which_data = "oecd", data_type = "time-series")
# Valitaa pari muutama muuttuja
oecd_subset <- oecd %>%
select(cname,year,contains("hdi")) %>%
# poistetaan na-sisältävät rivit
na.omit()
dim(oecd_subset)## [1] 993 3head(oecd_subset)## cname year undp_hdi
## 45 Australia 1990 0.866
## 46 Australia 1991 0.867
## 47 Australia 1992 0.868
## 48 Australia 1993 0.872
## 49 Australia 1994 0.875
## 50 Australia 1995 0.883# suomidata
d_fi <- oecd_subset %>% filter(cname == "Finland")
head(d_fi)## cname year undp_hdi
## 1 Finland 1990 0.784
## 2 Finland 1991 0.787
## 3 Finland 1992 0.795
## 4 Finland 1993 0.799
## 5 Finland 1994 0.809
## 6 Finland 1995 0.816# ruotsidata
d_se <- oecd_subset %>% filter(cname == "Sweden")
head(d_se)## cname year undp_hdi
## 1 Sweden 1990 0.816
## 2 Sweden 1991 0.818
## 3 Sweden 1992 0.821
## 4 Sweden 1993 0.840
## 5 Sweden 1994 0.849
## 6 Sweden 1995 0.856# norjadata
d_no <- oecd_subset %>% filter(cname == "Norway")
head(d_no)## cname year undp_hdi
## 1 Norway 1990 0.850
## 2 Norway 1991 0.856
## 3 Norway 1992 0.862
## 4 Norway 1993 0.870
## 5 Norway 1994 0.885
## 6 Norway 1995 0.883Pinoa kolme dataa
d_skand <- bind_rows(d_fi,
d_se,
d_no)
sample_n(tbl = d_skand, size = 10)## cname year undp_hdi
## 1 Finland 2010 0.903
## 2 Sweden 1991 0.818
## 3 Finland 2002 0.867
## 4 Sweden 2016 0.932
## 5 Finland 2008 0.904
## 6 Norway 1998 0.906
## 7 Sweden 1992 0.821
## 8 Sweden 2002 0.902
## 9 Finland 2009 0.899
## 10 Finland 1995 0.816Sarakkeiden liittäminen rinnakkain dplyr::bind_cols()
oecd <- read_qog(which_data = "oecd", data_type = "time-series", year = 2019)
oecd <- as_tibble(oecd)
# Valitaa pari muutama muuttuja
fi_who <- oecd %>% select(cname,year,contains("who_")) %>% filter(cname == "Finland", year > 1990)
fi_fao <- oecd %>% select(cname,year,contains("fao_")) %>% filter(cname == "Finland", year > 1990)
bind_cols(fi_who,fi_fao) %>% slice(1:10)## # A tibble: 10 x 34
## cname year who_alcohol0009 who_alcohol8099 who_dwrur who_dwtot
## <chr> <int> <dbl> <dbl> <int> <int>
## 1 Finl… 1991 NA 9.22 NA NA
## 2 Finl… 1992 NA 8.88 NA NA
## 3 Finl… 1993 NA 8.39 NA NA
## 4 Finl… 1994 NA 8.16 NA NA
## 5 Finl… 1995 NA 8.31 NA NA
## 6 Finl… 1996 NA 8.24 NA NA
## 7 Finl… 1997 NA 8.56 NA NA
## 8 Finl… 1998 NA 8.60 NA NA
## 9 Finl… 1999 NA 8.62 NA NA
## 10 Finl… 2000 8.59 NA 100 100
## # … with 28 more variables: who_dwurb <int>, who_infmort <dbl>,
## # who_lef <dbl>, who_lem <dbl>, who_let <dbl>, who_mrf <int>,
## # who_mrm <int>, who_mrt <int>, who_neomort <dbl>, who_sanitrur <int>,
## # who_sanittot <int>, who_saniturb <int>, who_ufivemort <dbl>,
## # cname1 <chr>, year1 <int>, fao_luagr <dbl>, fao_luagrara <dbl>,
## # fao_luagrcrop <dbl>, fao_luagrirreq <dbl>, fao_luagrorg <dbl>,
## # fao_luagrpas <dbl>, fao_luagrpcrop <dbl>, fao_lucrop <dbl>,
## # fao_luforest <dbl>, fao_luforplant <dbl>, fao_luforprim <dbl>,
## # fao_luforreg <dbl>, fao_lupas <dbl>Datojen yhdistäminen avainmuuttujilla dplyr::join
library(dplyr)
filmit <- dplyr::starwars %>%
select(name,films) %>%
unnest() %>%
filter(grepl("^The", films))
filmit## # A tibble: 61 x 2
## name films
## <chr> <chr>
## 1 Luke Skywalker The Empire Strikes Back
## 2 Luke Skywalker The Force Awakens
## 3 C-3PO The Phantom Menace
## 4 C-3PO The Empire Strikes Back
## 5 R2-D2 The Phantom Menace
## 6 R2-D2 The Empire Strikes Back
## 7 R2-D2 The Force Awakens
## 8 Darth Vader The Empire Strikes Back
## 9 Leia Organa The Empire Strikes Back
## 10 Leia Organa The Force Awakens
## # … with 51 more rowsajoneuvot <- dplyr::starwars %>%
select(name,vehicles) %>%
unnest()
ajoneuvot## # A tibble: 13 x 2
## name vehicles
## <chr> <chr>
## 1 Luke Skywalker Snowspeeder
## 2 Luke Skywalker Imperial Speeder Bike
## 3 Leia Organa Imperial Speeder Bike
## 4 Obi-Wan Kenobi Tribubble bongo
## 5 Anakin Skywalker Zephyr-G swoop bike
## 6 Anakin Skywalker XJ-6 airspeeder
## 7 Chewbacca AT-ST
## 8 Wedge Antilles Snowspeeder
## 9 Qui-Gon Jinn Tribubble bongo
## 10 Darth Maul Sith speeder
## 11 Dooku Flitknot speeder
## 12 Zam Wesell Koro-2 Exodrive airspeeder
## 13 Grievous Tsmeu-6 personal wheel bikeihmiset <- dplyr::starwars %>%
select(name,height,mass,species) %>%
filter(species == "Human")
ihmiset## # A tibble: 35 x 4
## name height mass species
## <chr> <int> <dbl> <chr>
## 1 Luke Skywalker 172 77 Human
## 2 Darth Vader 202 136 Human
## 3 Leia Organa 150 49 Human
## 4 Owen Lars 178 120 Human
## 5 Beru Whitesun lars 165 75 Human
## 6 Biggs Darklighter 183 84 Human
## 7 Obi-Wan Kenobi 182 77 Human
## 8 Anakin Skywalker 188 84 Human
## 9 Wilhuff Tarkin 180 NA Human
## 10 Han Solo 180 80 Human
## # … with 25 more rowsleft_join(ihmiset, filmit, by = "name")## # A tibble: 40 x 5
## name height mass species films
## <chr> <int> <dbl> <chr> <chr>
## 1 Luke Skywalker 172 77 Human The Empire Strikes Back
## 2 Luke Skywalker 172 77 Human The Force Awakens
## 3 Darth Vader 202 136 Human The Empire Strikes Back
## 4 Leia Organa 150 49 Human The Empire Strikes Back
## 5 Leia Organa 150 49 Human The Force Awakens
## 6 Owen Lars 178 120 Human <NA>
## 7 Beru Whitesun lars 165 75 Human <NA>
## 8 Biggs Darklighter 183 84 Human <NA>
## 9 Obi-Wan Kenobi 182 77 Human The Phantom Menace
## 10 Obi-Wan Kenobi 182 77 Human The Empire Strikes Back
## # … with 30 more rowsright_join(ihmiset, filmit, by = "name")## # A tibble: 61 x 5
## name height mass species films
## <chr> <int> <dbl> <chr> <chr>
## 1 Luke Skywalker 172 77 Human The Empire Strikes Back
## 2 Luke Skywalker 172 77 Human The Force Awakens
## 3 C-3PO NA NA <NA> The Phantom Menace
## 4 C-3PO NA NA <NA> The Empire Strikes Back
## 5 R2-D2 NA NA <NA> The Phantom Menace
## 6 R2-D2 NA NA <NA> The Empire Strikes Back
## 7 R2-D2 NA NA <NA> The Force Awakens
## 8 Darth Vader 202 136 Human The Empire Strikes Back
## 9 Leia Organa 150 49 Human The Empire Strikes Back
## 10 Leia Organa 150 49 Human The Force Awakens
## # … with 51 more rowsinner_join(ihmiset, filmit, by = "name")## # A tibble: 24 x 5
## name height mass species films
## <chr> <int> <dbl> <chr> <chr>
## 1 Luke Skywalker 172 77 Human The Empire Strikes Back
## 2 Luke Skywalker 172 77 Human The Force Awakens
## 3 Darth Vader 202 136 Human The Empire Strikes Back
## 4 Leia Organa 150 49 Human The Empire Strikes Back
## 5 Leia Organa 150 49 Human The Force Awakens
## 6 Obi-Wan Kenobi 182 77 Human The Phantom Menace
## 7 Obi-Wan Kenobi 182 77 Human The Empire Strikes Back
## 8 Anakin Skywalker 188 84 Human The Phantom Menace
## 9 Han Solo 180 80 Human The Empire Strikes Back
## 10 Han Solo 180 80 Human The Force Awakens
## # … with 14 more rowsfull_join(ihmiset, filmit, by = "name")## # A tibble: 77 x 5
## name height mass species films
## <chr> <int> <dbl> <chr> <chr>
## 1 Luke Skywalker 172 77 Human The Empire Strikes Back
## 2 Luke Skywalker 172 77 Human The Force Awakens
## 3 Darth Vader 202 136 Human The Empire Strikes Back
## 4 Leia Organa 150 49 Human The Empire Strikes Back
## 5 Leia Organa 150 49 Human The Force Awakens
## 6 Owen Lars 178 120 Human <NA>
## 7 Beru Whitesun lars 165 75 Human <NA>
## 8 Biggs Darklighter 183 84 Human <NA>
## 9 Obi-Wan Kenobi 182 77 Human The Phantom Menace
## 10 Obi-Wan Kenobi 182 77 Human The Empire Strikes Back
## # … with 67 more rowssemi_join(filmit, ihmiset, by = "name")## # A tibble: 24 x 2
## name films
## <chr> <chr>
## 1 Luke Skywalker The Empire Strikes Back
## 2 Luke Skywalker The Force Awakens
## 3 Darth Vader The Empire Strikes Back
## 4 Leia Organa The Empire Strikes Back
## 5 Leia Organa The Force Awakens
## 6 Obi-Wan Kenobi The Phantom Menace
## 7 Obi-Wan Kenobi The Empire Strikes Back
## 8 Anakin Skywalker The Phantom Menace
## 9 Han Solo The Empire Strikes Back
## 10 Han Solo The Force Awakens
## # … with 14 more rowsanti_join(filmit, ihmiset, by = "name")## # A tibble: 37 x 2
## name films
## <chr> <chr>
## 1 C-3PO The Phantom Menace
## 2 C-3PO The Empire Strikes Back
## 3 R2-D2 The Phantom Menace
## 4 R2-D2 The Empire Strikes Back
## 5 R2-D2 The Force Awakens
## 6 Chewbacca The Empire Strikes Back
## 7 Chewbacca The Force Awakens
## 8 Jabba Desilijic Tiure The Phantom Menace
## 9 Yoda The Phantom Menace
## 10 Yoda The Empire Strikes Back
## # … with 27 more rowsLisäverbejä joineihin
dplyr::union
dplyr::intersect
dplyr::setdifLuento 4: Erilaiset visualisoinnit ja analyysiprosessin kokonaisuus
Datojen vieminen toisiin ohjelmiin
Lyhyenä kertauksena vielä miten voit tuoda datoja R:ään ja viedä datojen R:stä toisiin ohjelmiin. Tähän mennessä olemme tuoneet datoja haven-paketin funktioilla, readr::read_csv()-funktiolla sekä readxl::read_excel()-funktiolla. Nämä paketit tarjoavat kirjoittamiseen vastaavat funktiot, jotka havenissa ovat haven::write_dta() jne. readr-paketilla kirjoitat .csv-datan readr::write_csv()-funktiolla. Excel-tiedoston voit kirjoittaa funktiolla writexl::write_xlsx().
Yleensä varmin tapa siirtää data R:stä SAS:iin, Stata:an tai SPSS:ään on a) tallentaa data ensin .csv-muotoon ja lukea se sitten ko. ohjelmaan ns. tekstinä. Kussakin ohjelmassa on oma tapansa tekstidatojen tuomiselle ja manuaalit/google on tässä hyvä apu.
Mikäli näiden eri pakettien muistaminen tuntuu työläältä kannattaa tututustua myös pakettiin rio: A Swiss-Army Knife for Data I/O. Paketissa on käytänössä kaksi funktiota import() ja export() ja funktiot arvaa luettavan/kirjoitettavan tiedoston päätteestä mitä pakettia käyttää. Eli jos haluat lukea vaikkapa Statan formaattia tai .csv-tiedoston, voit kirjoittaa vain näin:
ess <- rio::import(file = "./datasetit/ESS8e01.stata/ESS8e01.dta")
oecd <- rio::import(file = "./datasetit/qog_oecd_ts_jan18.csv")Jos taas haluat tallentaa dplyr::starwars-datan exceliksi ja csv:ksi, voit tehdä sen
sw <- dplyr::starwars
rio::export(x = sw, file = "./datasetit/starwars.xlsx") # excel
rio::export(x = sw, file = "./datasetit/starwars.csv") # csvVisualisoinnit
Seuraavassa käydään läpi aluksi ggplot2-kuvien ulkoasun räätälöimistä ja lopuksi kuvien tallentamista levylle.
Kuvien ulkoasun räätäloiminen
Tässä on yksinkertainen demo yhden kuvan räätälöimisestä. Ennen sen läpikäymistä tutustu kurssikurjoita lukuun 8 Refine your plots!
Likipitäen kaikia ggplot2-kuvien elementtejä on mahdollista muokata. Helpoin tapa päästä alkuun on ottaa joku valmis teema ja viilata sitä omien/kollegoiden/lehden vaatimuksien mukaiseksi.
Teen seuraavassa tuttuun starwars-dataan pari uutta muuttujaa ja ehostan kuvaa askel askeleelta.
library(tidyverse)
# luodaan data ja poistetaan Jabba
sw <- dplyr::starwars %>% filter(name != "Jabba Desilijic Tiure") %>%
# tehdään syntymävuosiluokkamuuttuja
mutate(birth_year_class = case_when(
birth_year <= 34 ~ "the old",
birth_year > 34 & birth_year <= 70 ~ "the middle aged",
birth_year > 70 ~ "the young",
TRUE ~ "not born"
),
# Tehdään muuttujasta faktori!
birth_year_class = factor(birth_year_class, levels = c("the young",
"the middle aged",
"the old",
"not born")),
# tehdään mys painoindeksi
bmi = mass / (height/100)^2) %>%
arrange(bmi) %>%
slice(1:25)Askel 1: Peruskuva
Tehdään hajontakuvio jossa kaikki uudet muuttujat näytillä!
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name))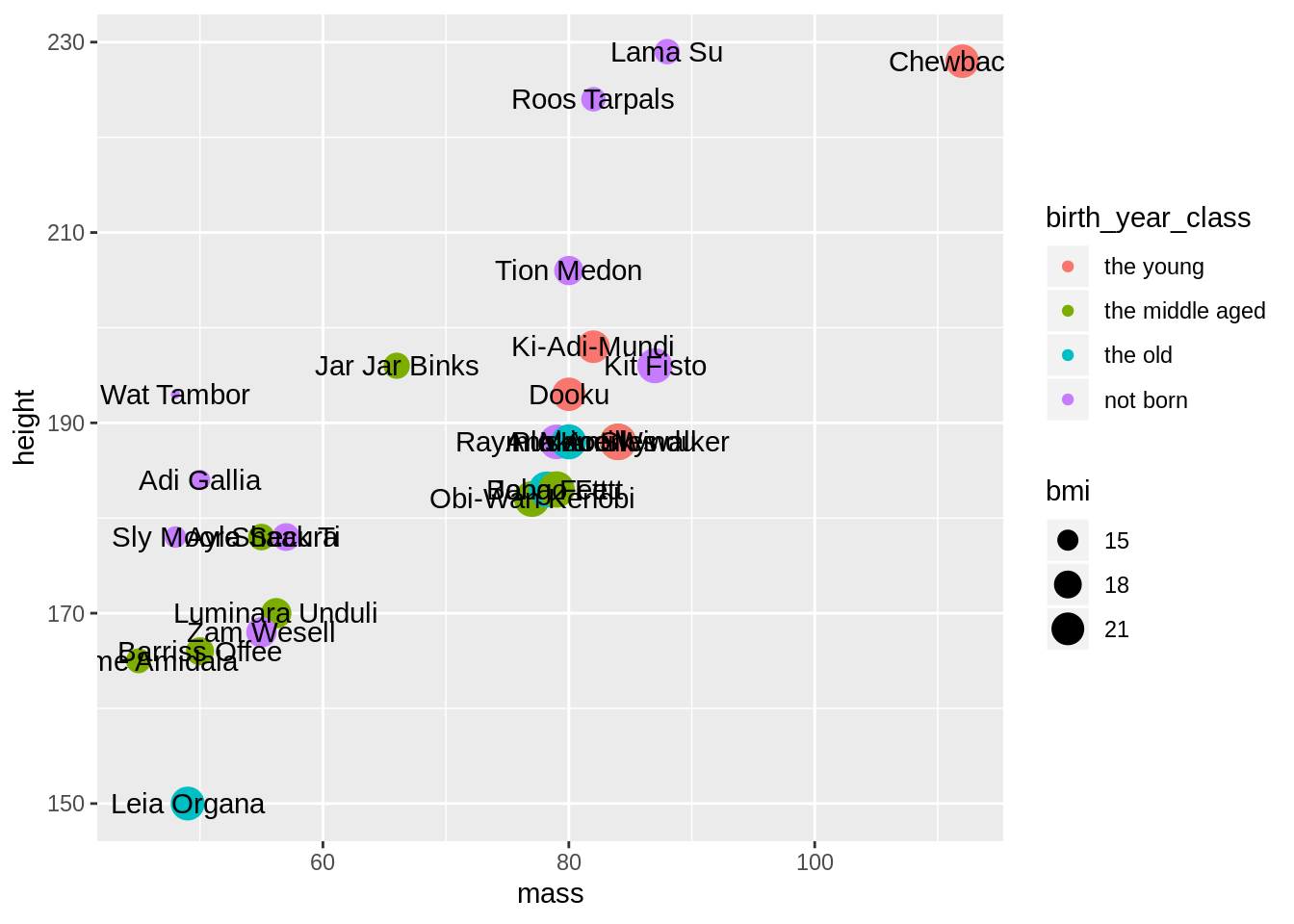
Askel 2: Valmiit teemat
ggplot2-paketissa on mukana 10 teemaa, jotka saat käyttöön lisäämällä theme_teemanimi()-kerroksen kuvaan. Voit antaa suoraan kullekin teemalle argumenteiksi base_size eli teeman fonttikoon sekä base_family eli teeman fontin. Tässä vaiheessa lisään vain pelkän teeman. Otsikot ovat jo tuttu asia ja lisään tässä vaiheessa kuvaan myös otsikkokerroksen funktiolla labs()
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name)) +
# Teema
theme_minimal() +
# Kuvan otsikot
labs(title = "Starwars otusten kehonkoostumus ja elinvuodet",
subtitle = "Esimerkkikuva ggplot2:n perusominaisuuksista",
size = "Painoindeksi",
color = "Luokiteltu ikä",
x = "Paino (kg)",
y = "Pituus (cm)",
caption = "data: dplyr::starwars")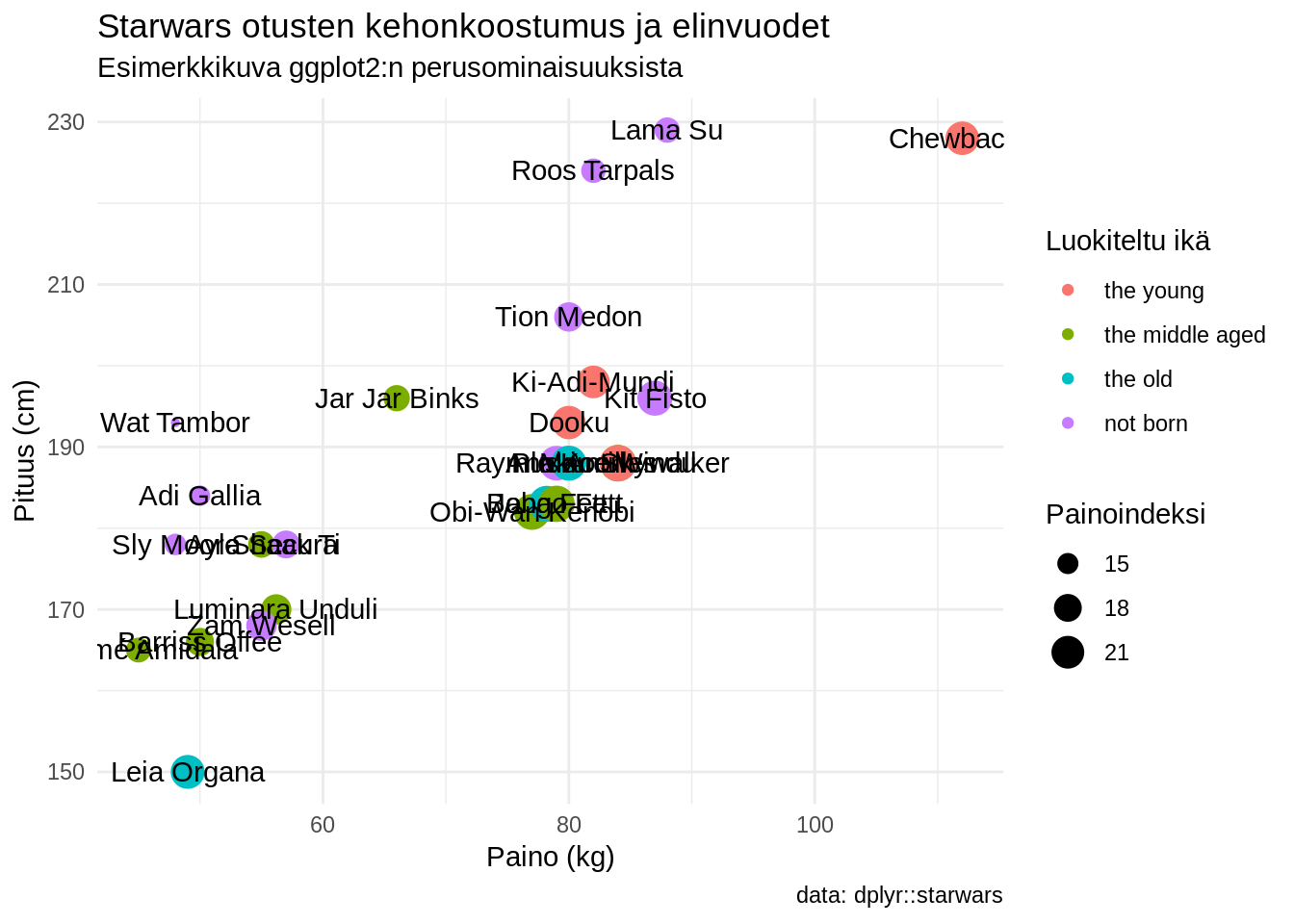
Askel 3: Värit
Riittävän hyvän väripaletin löytäminen kulloiseenkin tarkoitukseen on usein aikaavievä ja hermoja raastava vaihe. R:ssä on saatavilla muutama sata väriä jotka listattu mm. dokumenttiin Colors in R ja joita voi kutsua suoraan värin nimellä. ggplot2:ssa voit luoda omia väripaletteja funktioilla scale_color_manual() ja scale_fill_manual(). Funktiolla scale_fill_brewer(palette = "paletinnimi")/scale_color_brewer(palette = "paletinnimi") voit lisätä valmiita ColorBrewer-paletteja Alla kuvaan on lisätty manuaalinen väri palloille.
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name)) +
# Teema
theme_minimal() +
# Kuvan otsikot
labs(title = "Starwars otusten kehonkoostumus ja elinvuodet",
subtitle = "Esimerkkikuva ggplot2:n perusominaisuuksista",
size = "Painoindeksi",
color = "Luokiteltu ikä",
x = "Paino (kg)",
y = "Pituus (cm)",
caption = "data: dplyr::starwars") +
# lisätään oma väripaletti
scale_color_manual(values = c("royalblue", "springgreen", "red", "darkmagenta"))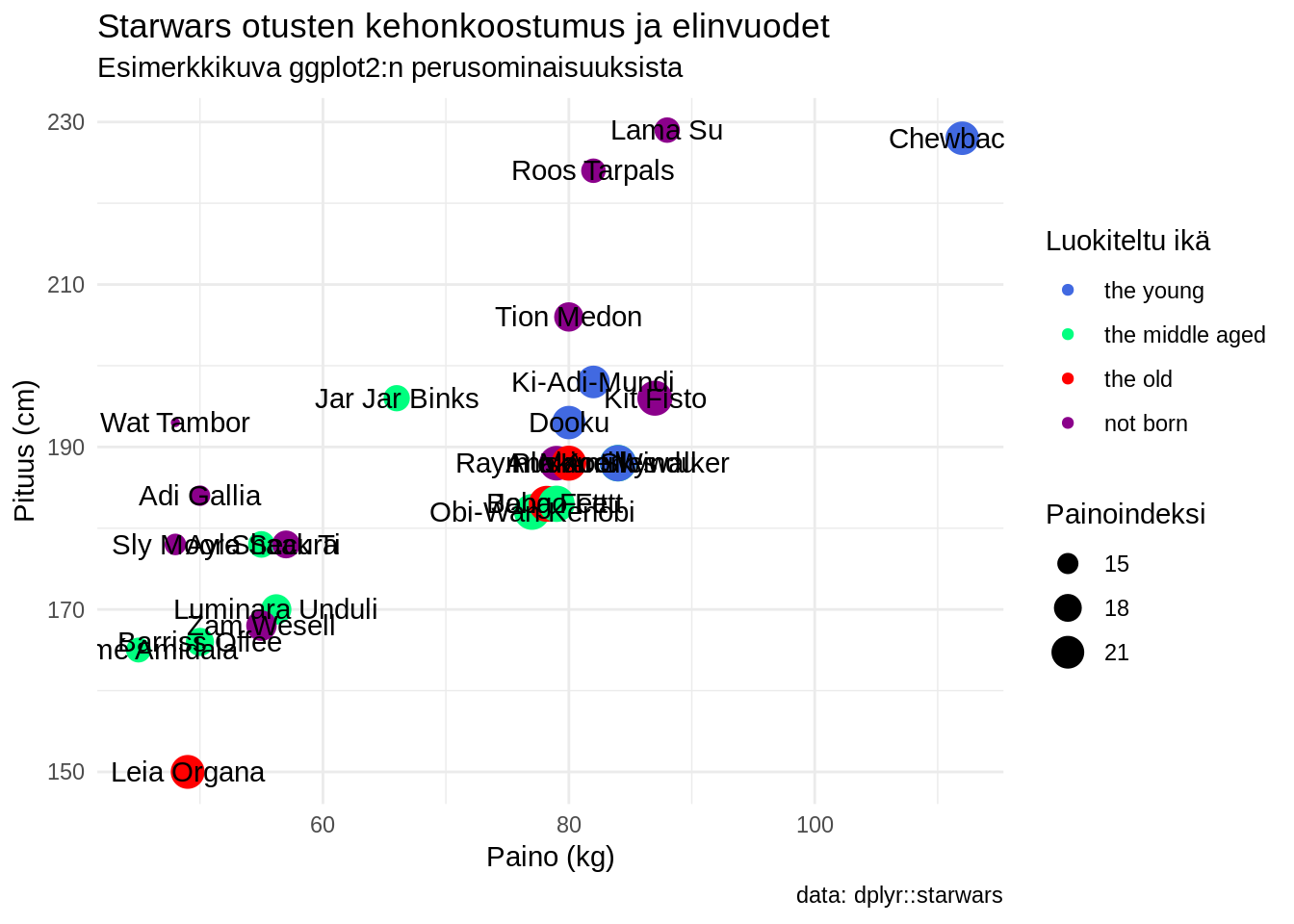
R:ään on olemassa useita valmiita väripaletteja sisältäviä paketteja. Emil Hvitfeldtin Comprehensive list of color palettes in r lienee kattavin yhteenveto paleteista. Itse käytän usein paketteja ggsci ja viridis. Kokeile vaikka ggsci::scale_color_futurama()
Askel 4: Fontit
Voit käyttää koneellesi asennettuja fontteja R:ssä extrafont-paketin avulla. Kun käytät paketettia ensimmäisen kerran, aja seuraavat komennot:
install.packages("extrafont") # asentaa paketin
library(extrafont) # lataa paketin
font_import() # tekee koneella olevista fonteista tietokannan paketin asennushakemistoonKun otat fontit myöhemmin käyttöön riittää kun ennen kuvan piirtoa aja komenot
library(extrafont)
loadfonts(quiet = TRUE) # lataa fontit asennushakemiston tietokannastaSaat selville saatavilla olevat fontit komennolla extrafont::fonts(). Alla olevassa kuvassa olen antanut theme_minimal() funktiolle argumentiksi erään koneelleni asennetun fontin nimen Ubuntu. Fontin voi asentaa mm. google web fonts -palvelusta, mikäli sinulla on pääkäyttäjän oikeudet koneelle.
library(extrafont)
loadfonts(quiet = TRUE)
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name)) +
# Teema
theme_minimal(base_family = "Ubuntu") +
# Kuvan otsikot
labs(title = "Starwars otusten kehonkoostumus ja elinvuodet",
subtitle = "Esimerkkikuva ggplot2:n perusominaisuuksista",
size = "Painoindeksi",
color = "Luokiteltu ikä",
x = "Paino (kg)",
y = "Pituus (cm)",
caption = "data: dplyr::starwars") +
# lisätään oma väripaletti
scale_color_manual(values = c("royalblue", "springgreen", "red", "darkmagenta"))
Askel 5: Teemojen kustomointi
Kuvan eri komponenteja muokataan funktion theme() avulla. Virallisen dokumentaation sivu Modify components of a theme kertoo kunkin muokattavan komponentin tyypin ja muokkausmahdollisuudet. Seuraavassa siiräämme selitteen alhaalle ja tasaamme akselin otsikot lähelle origoa.
library(extrafont)
loadfonts(quiet = TRUE)
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name)) +
# Teema
theme_minimal(base_family = "Ubuntu") +
# Kuvan otsikot
labs(title = "Starwars otusten kehonkoostumus ja elinvuodet",
subtitle = "Esimerkkikuva ggplot2:n perusominaisuuksista",
size = "Painoindeksi",
color = "Luokiteltu ikä",
x = "Paino (kg)",
y = "Pituus (cm)",
caption = "data: dplyr::starwars") +
# lisätään oma väripaletti
scale_color_manual(values = c("royalblue", "springgreen", "red", "darkmagenta")) +
# Teeman kustomointi
theme(legend.position = "bottom",
axis.title = element_text(hjust = 0))
Askel 6: Tekstilabeleiden kustomointi
Tässä hajontakuviossa olen lisännyt pisteille myös kunkin hahmon nimen. Teeman fontin muokkaaminen ei muuta labeleiden fontteja, vaan se on tehtävä käsin. Seuraavassa muokkaa labeleiden fonttia ja nostan kutakin labelia hieman pisteen yläpuolelle. Huomaa että teen muutokset ~koodin puoliväliin
library(extrafont)
loadfonts(quiet = TRUE)
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name), family = "Ubuntu", fontface = "bold", size = 3, nudge_y = 4) +
# Teema
theme_minimal(base_family = "Ubuntu") +
# Kuvan otsikot
labs(title = "Starwars otusten kehonkoostumus ja elinvuodet",
subtitle = "Esimerkkikuva ggplot2:n perusominaisuuksista",
size = "Painoindeksi",
color = "Luokiteltu ikä",
x = "Paino (kg)",
y = "Pituus (cm)",
caption = "data: dplyr::starwars") +
# lisätään oma väripaletti
scale_color_manual(values = c("royalblue", "springgreen", "red", "darkmagenta")) +
# Teeman kustomointi
theme(legend.position = "bottom",
axis.title = element_text(hjust = 0))
Askel 7: Päällekkäisen piirtämisen (overplotting) hallinta
Etenkin hajontakuvioissa, pisteet ja etenkin labelit menevät usein päällekkäin. Pisteiden päällekkäisyyttä voi ggplot2:ssa hoitaa “hajottamalla” pisteiden sijaintia käyttämällä geom_point():n sijaan geom_jitter():ia. Tässä tapauksessa riittää pelkkä läpinäkyvyyden lisääminen alpha argumentilla. Labeleiden päällekkäisyys hoidetaan asentamalla uusi paketti ggrepel. Alla kuva jossa sekä geom_point() että geom_text() kerrokset muokattu.
library(extrafont)
loadfonts(quiet = TRUE)
# peruskerrokseen annetaan data sekä x ja y muuttujat
ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi), alpha = .6) +
# tekstille määritetään ainoastaan nimi
ggrepel::geom_text_repel(aes(label = name), family = "Ubuntu", fontface = "bold", size = 3, nudge_y = 4) +
# Teema
theme_minimal(base_family = "Ubuntu") +
# Kuvan otsikot
labs(title = "Starwars otusten kehonkoostumus ja elinvuodet",
subtitle = "Esimerkkikuva ggplot2:n perusominaisuuksista",
size = "Painoindeksi",
color = "Luokiteltu ikä",
x = "Paino (kg)",
y = "Pituus (cm)",
caption = "data: dplyr::starwars") +
# lisätään oma väripaletti
scale_color_manual(values = c("royalblue", "springgreen", "red", "darkmagenta")) +
# Teeman kustomointi
theme(legend.position = "bottom",
axis.title = element_text(hjust = 0))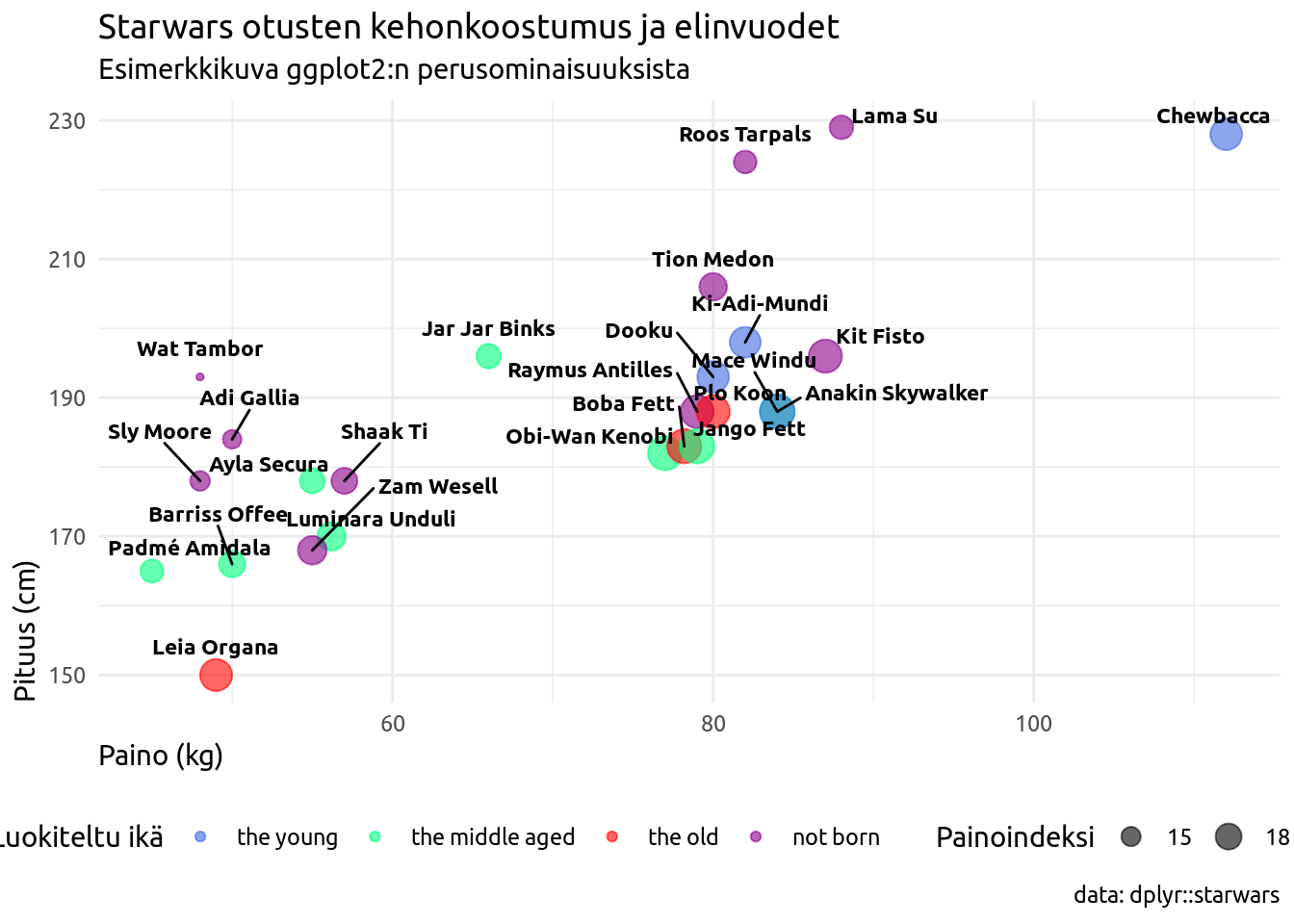
Lyhyt johdanto grafiikkaformaatteihin
Tilastografiikat tekemisessä on tärkeää tietää perusasiat eri grafiikkaformaateista. Grafiikkaformaattien kaksi päälinjaa ovat bittikarttagrafiikka ja vektorigrafiikka, joiden ominaisuudet tiivistetty ao. taulukkoon. Lue myös yo. lyhyet wikipedia-artikkelit mikäli ero ei ole aivan selvä.
Vektorigrafiikka vs. bittimappigrafiikka
| bittikartta (bitmap) | vektori | |
|---|---|---|
| tiedostopääte | .jpg, .png, .gif | .eps, .pdf, .svg, .ai |
| esimerkiksi | digikuva | googlen kartat |
| koostuu | miljoonista pikseleistä | pisteistä, viivoista ja polygoneista |
| tiedostokoko | suuri | pieni |
| muokkausohjelmisto | Gimp (Photoshop) | Inkscape (Illustrator) |
| sopii | verkoon, printtiin (korkearesoluutioisena) | printtiin, jälkikäsittelyyn, -svg-muodossa myös verkkoon |
Vektorigrafiikasta voi tehdä bittimappigrafiikkaa, mutta ei toisin päin #’ R:ssä tehtyjä kuvia voit tallentaa useisiin erilaisiin sekä vektori- että bittikarttaformaattiehin.
Staattinen vs. vuorovaikutteinen grafiikka
Bittikartta- ja vektorigrafiikka ovat ensisijaisesti ns. staattista grafiikka, jossa ei ole vuorovaikutteisia ominaisuuksia. “Tutkimusviestinnässä” perinteisen printattavan tutkimusraportin ohella yleistyy erilaiset verkkototeutukset kuten blogit tai verkkosovellukset. Kun sisältö on palvelimella ja lukeminen tapahtuu selaimella, niin kaikenlaiset verkkoteknologiat ovat käytettävissä. Verkkototeutusten kaksi päälinjaa, vuorovaikutteinen grafiikka & verkkosovellukset, on vedetty yhteen alla olevaan taulukkoon.
| grafiikan tyyppi | pros | cons |
|---|---|---|
| interaktiiviset kuviot | teknologian kehitys nopeaa | lyhyt elinkaari päivittyvien riippuvuuksien myötä |
| paljon vaihtoehtoja | hyvin sekava skene | |
| tarvitsee verkkoyhteydet | ||
| ei voi printata! | ||
| verkkosovellukset | hyvin joustavia | hostaus ja ylläpito |
| pystyy kaikkeen mihin R & linux | vaatii paljon räätälöintia |
Vuorovaikutteiset kuviot
Helpoin tapa tehdä ggplot2-kuvista vuorovaikutteisia on asentaa plotly()-paketti.
kuvaobjekti <- ggplot(data = sw, aes(x = mass, y = height)) +
# pisteille määritetään lisäksi väri ja koko
geom_point(aes(color = birth_year_class, size = bmi)) +
# tekstille määritetään ainoastaan nimi
geom_text(aes(label = name))
plotly::ggplotly(kuvaobjekti)Lisää tietoa erilaisista vuorovaikutteisen grafiikan paketeista löytyy sivulta htmlwidgets - Showcase.
verkkosovellukset
shiny-paketin avulla voit tehdä vuorovaikutteisia analyysisovelluksia nettiselaimeen. Sovellukset ovat käteviä uuteen dataan tutustuttaessa, mutta shinylla voi tehdä myös monimutkaisempia sovelluksia ja niitä voi jakaa joko shinyapps.io:n tai omalle palvelimelle asennettavan shiny server:in kautta.
Tein pienen demon, jonka voit ladata itsellesi seuraavasti
# 1. päivitä kurssin paketti
download.file("http://courses.markuskainu.fi/utur2018/harjoitukset/app.R", "app.R")
# 3. avaa sovellus muokattavaksi
file.edit("./app.R")
# 4. paina muokkausikkunan oikeasta yläkulmasta "Run App" -nappulaa!Tarkastele koodia, muokkaa ja kokeile tehdä siihen uusia ominaisuuksia!
Kuvien tallentaminen levylle
Rstudiossa voit valita Plots-paneelista Export ja tallentaa kuvan Save as image dialogista kuuteen eri formaattiin:
- PNG - bittikartta-formaatti - tukee läpinäkyvyyttä - varma valinta verkkoon
- JPEG/JPG) - bittikartta-formaatti - pakattavissa pieneen tilaan - varma valinta verkkoon
- TIFF - bittikartta-formaatti - pakkaamaton, korkearesoluutioisena printtiin
- BMP - bittikartta-formaatti - perinteinen pakkaamaton formaatti. katoamassa.
- SVG - scalable vector graphics - avoin vektorigrafiikkamuoto. Uudet selaimet tukevat. Inkscape-grafiikkaohjelman oletusmuoto
- EPS - Encapsulated PostScript -
sekä pdf-formaattiin kohdasta “Save as PDF”.
pdf-formaatti on nykyisellään parhaiten yhteensopiva vektorigrafiikan muoto. Suurin haaste on eri fonttien käyttö
ggplot2-kuvien tallentaminen eri formaatteihin
ggplot2-paketilla tehtyjä kuvia voi tallentaa Rstudio-käyttöliittymästä käsin samoin kuin kaikkia muitakin formaatteja. Tallentaminen on kuitenkin näppärämpää kirjoittaa kuvion koodin yhteyteen ´ggsave()-funktiota käyttäen. ´ggsave()-funktio ymmärtää kuvion nimen päätteestä, mihin formaattiin kuva tallennetaan. Jos teet kuvion alla olevalla koodilla
kuvaobjekti <- ggplot(data=cars, aes(x=speed,y=dist)) + geom_point()
Voit tallentaa sen eri formaatteihin koodilla
ggsave(plot=kuvaobjekti, filename = "kuva.png")- bittimappi pngggsave(plot=kuvaobjekti, filename = "kuva.pdf")- vektori pdfggsave(plot=kuvaobjekti, filename = "kuva.svg")- vektori svg
Lisäparametreinä ´ggsave()`-funktioon voi laittaa mm. seuraavat
filenameFile name to create on disk.plotPlot to save, defaults to last plot displayed.deviceDevice to use (function or any of the recognized extensions, e.g. “pdf”). By default, extracted from filename extension. ggsave currently recognises eps/ps, tex (pictex), pdf, jpeg, tiff, png, bmp, svg and wmf (windows only).pathPath to save plot to (combined with filename).scaleMultiplicative scaling factor.width,heightPlot dimensions, defaults to size of current graphics device.unitsUnits for width and height when specified explicitly (in, cm, or mm)dpiResolution used for raster outputs.limitsizeWhen TRUE (the default), ggsave will not save images larger than 50x50 inches, to prevent the common error of specifying dimensions in pixels.
Mikäli et ole vielä luonut kansiota kotitehtava4/kuviot luo se komennolla dir.create(path="./kotitehtava4/kuviot", recursive=TRUE, showWarnings = FALSE)
Piirrä kuvat koodilla kuva <- ggplot(data=cars, aes(x=speed,y=dist)) + geom_point()` ja kirjoita miten se tallennetaan ko. kansioon png, svg, tiff, jpg, eps, pdf ja bmp muodoissa.
Avaan kaikki kuvat koneellasi ja tarkastele niitä!
Kartat
Perinteisen tilastollisen ohjelmsiston lisäksi R:ssä on erinomaiset spatiaalisen datan käsittelyn ja analysoinnin valmiudet. Internet on pullollaan erilaisia tutoriaaleja karttojen tekemisestä R:llä, mutta kokonaisuus on liian laaja tällä kurssilla käsiteltäväksi linkitän tähän vain pari hyvää resurssia joilla pääsee alkuun:
- Kurssikirjasta luku 7 Draw maps
- Euroopan maiden karttoja Eurostatin datoilla
- Pari omaa viimeistellympää demoa Eurostatin datoista Eurostat (regional) maps using R and eurostat-package
Analyysiprosessin kokonaisuus
Data-analyysiprosessin kokonaisuuden hallintaan ei ole olemassa yhtä oikeaa vaihtoehtoa. Oikea projektin jäsentämisen ja tiedonhallinnan malli riippuu aina tehtävästä ja tekijöistä. Kaksi välinettä 1) Rstudion projektit ja 2) Rmarkdown ovat kuitenkin ehdottoman hyödyllisiä. Projekteista on kurssin aikana jo puhuttu, samoin Rmarkdownista, mutta tutustu jälkimmäiseen paremmin vielä alla olevan luennon pohjalta.
Luennon ohella perehdy myös tähän artikkeliin Wilson G, Bryan J, Cranston K, Kitzes J, Nederbragt L, Teal TK (2017) Good enough practices in scientific computing. PLoS Comput Biol 13(6): e1005510.. Siinä käydään läpi hyvän data-analyysin peruskäsitteitä ja ehdotetaan myös yhtä melko hyvää tiedonhallinnan mallia.
Rmarkdown tilastollisten raporttien kirjoittamisessa
Rmarkdownin laajennokset
2017-2019 Markus Kainu.

Tämä teos on lisensoitu Creative Commons Nimeä 4.0 Kansainvälinen -lisenssillä.